用于过继免疫疗法的组合物和方法
阅读说明:本技术 用于过继免疫疗法的组合物和方法 (Compositions and methods for adoptive immunotherapy ) 是由 杨文� 于 2020-07-22 设计创作,主要内容包括:本发明涉及赋予和/或增加由细胞免疫疗法介导的免疫应答的药剂、组合物和方法。(The present invention relates to agents, compositions and methods for conferring and/or increasing immune responses mediated by cellular immunotherapy.)
相关申请的交叉引用
本申请要求2019年07月23日提交的美国临时申请号62/877,331和2020年06月25日提交的美国临时申请号63/044,059的优先权。该申请的内容通过引用整体并入本文。
发明领域
本发明涉及重组抗原受体及其用途。经过工程化改造以表达此类抗原受体的T细胞可用于治疗以表达一种或多种与所述抗原受体结合的抗原为特征的疾病。
背景技术
过继免疫疗法涉及向患者施用免疫效应细胞以产生治疗作用。嵌合抗原受体(CAR)T细胞的出现为改善过继免疫疗法提供了有用的工具。为此,使用基因工程将定义特异性的抗原靶向受体插入T细胞,这极大地扩展了过继免疫疗法的能力。CAR是一种类型的抗原靶向受体,其由与胞外抗原结合域融合的胞内T细胞信号传导域组成。基于抗体的CAR直接识别细胞表面抗原,而不依赖于主要组织相容性复合物(MHC)介导的呈递,从而允许在所有患者中使用对任何给定抗原具有特异性的单个受体构建体。最初,CAR将抗原识别域融合至T细胞受体(TCR)复合物的CD3ζ(CD3ζ)活化链。随后的CAR迭代包括与CD3ζ串联的次级共刺激信号,包括来自CD28的胞内域或多种TNF受体家族分子,如4-1BB(CD137)和OX40(CD134)。除了CD3ζ以外,第三代受体包括两种不同的共刺激信号,最常见的来自CD28和4-1BB。第二代和第三代基于抗体的CAR在体外和体内均改善了抗肿瘤有效性。
转基因T细胞受体(tTCR)和基于TCR的CAR(TCR-CAR)与基于抗体的CAR不同,其主要在于tTCR和基于TCR的CAR以MHC(HLA)限制性方式结合至抗原。因此,tTCR和基于TCR的CAR极大地扩展了可能的靶点列表。此前已经描述了以两种单独的病毒载体形式建立的包含人CD28和CD3ζ(CD3Z)的胞质域或人CD28和CD3ε(CD3E)的胞质域的第二代TCR-CAR(Govers等,Journal of Immunology,2014,193:5315–5326),以及此前在美国专利号9,206,440和Im EJ等,Recombination–deletion between homologous cassettes inretrovirus is suppressed via a strategy of degenerate codonsubstitution.Molecular Therapy—Methods&Clinical Development(2014)Articlenumber:14022中描述的以单一病毒载体形式建立的包含人CD28和CD3ζ的胞质域的第二代TCR-CAR。此外,此前还描述了以单一病毒载体形式建立的包含单链TCR和人CD28和CD3ζ的胞质域的第二代TCR-CAR(Walseng等,A TCR-based Chimeric Antigen Receptor.SciRep.2017Sep 6;7(1):10713)。除了CD3ζ以外,第三代受体包括两种不同的共刺激信号,最常见的来自CD28和4-1BB。第二代和第三代基于抗体的CAR在体外和体内改善了抗肿瘤有效性。
与第二代或第三代基于抗体的CAR不同的是,tTCR和基于TCR的CAR面临各种障碍,如为了有效的tTCR或基于TCR的CAR表达和活性而失去稳定性。尽管tTCR本身在其自身分子中缺乏共刺激信号传导,但目前报道的基于TCR的CAR缺乏足够的共刺激信号传导元件(如4-1BB)或缺乏针对最佳细胞表面表达盒CAR介导的T细胞活性的最佳设计。对新的基于TCR的CAR设计以及提供具有增强功能的细胞的过继疗法存在需求。
发明内容
本公开内容在多个方面解决了上述需求。在一个方面中,本公开内容提供了一种抗原受体,其包含(I)第一多肽链,所述第一多肽链包含胞外域、跨膜(TM或Tm)域(TMD)和胞质域(Cyt或cyt),所述胞外域包含TCRβ链或其抗原结合片段;和(II)第二多肽链,所述第二多肽链包含胞外域(Ec)、跨膜域和胞质域,所述胞外域包含TCRα链或其抗原结合片段。所述TCRβ链和所述TCRα链形成抗原结合位点。在实施方式中,所述第一多肽链在所述跨膜域和所述胞质域的一个或多个中基本上不同于所述第二多肽链。
所述第一多肽链的所述胞质域或所述第二多肽链的所述胞质域包含(a)0、1或2个拷贝的人4-1BB或其片段的胞质域,或(b)0、1或2个拷贝的人CD3ζ(CD3Z)或其片段的胞质域,或(c)0、1或2个拷贝的人CD3ε(CD3E)或其片段的胞质域,或(d)0、1或2个拷贝的人CD28或其片段的胞质域的。
在所述抗原受体中,所述第一多肽链的所述跨膜域或所述第二多肽链的所述跨膜域可以包含选自以下的一者:CD8的跨膜域和CD28的跨膜域。
在一些实施方式中,所述抗原受体包含1或2个拷贝的人4-1BB的所述胞质域。此类抗原受体的实例包括由本文所述的载体NT 4、5、6、21、22、23、24、25和27编码的那些。在一些实例中,所述抗原受体可以包含1或2个拷贝的人CD3ζ的所述胞质域。在其他实例中,所述抗原受体可以包含1或2个拷贝的人CD28的所述胞质域。实例包括由载体NT 6、21、22、23、24、25和27编码的那些。
在一个实施方式中,所述抗原受体仅包含1个拷贝的人4-1BB的所述胞质域、1个拷贝的人CD28的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。所述抗原受体还可以仅包含1个拷贝的CD28的所述跨膜域和1个拷贝的CD8的所述跨膜域。在一个实例中(如由NT22编码的),所述第一多肽链可以包含TCRβ链的所述胞外域、1个拷贝的所述CD28跨膜域、1个拷贝的人CD28的所述胞质域和1个拷贝的人CD3ζ的所述胞质域,而所述第二多肽链可以包含TCRα链的所述胞外域、1个拷贝的所述CD8的跨膜域和1个拷贝的人4-1BB的所述胞质域。
在一个实施方式中,所述抗原受体包含2个拷贝的人4-1BB的所述胞质域、2个拷贝的人CD28的所述胞质域和2个拷贝的人CD3ζ的所述胞质域。例如,两条链的每一个可以包含人4-1BB的所述胞质域、人CD28的所述胞质域和人CD3ζ的所述胞质域的每一者的1个拷贝(例如,由NT6编码)。或者,一条多肽链(例如,所述第一多肽链)可以包含2个拷贝的人CD28的所述胞质域,而另一条(例如,所述第二多肽链)可以包含2个拷贝的人4-1BB的所述胞质域。实例包括由载体NT25编码的。
在所述抗原受体的一个实施方式中,两条链中的一条具有人CD3ζ的所述胞质域。例如,所述第一多肽链可以包含1个拷贝的人CD28的所述跨膜域、1个拷贝的人CD28的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。所述第二多肽链可以包含1个拷贝的人CD8的所述跨膜域和1个拷贝的人4-1BB的所述胞质域,但不含人CD3ζ的胞质域。实例包括由NT22编码的。
在一个实施方式中,所述第一多肽链和所述第二多肽链具有不同的共刺激域。例如,所述第一多肽链可以包含1个拷贝的人CD8的所述胞质域、1个拷贝的人CD28的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。所述第二多肽链可以包含1个拷贝的人CD8的所述跨膜域、1个拷贝的人4-1BB的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。实例包括由NT24编码的。
在一个实施方式中,所述第一多肽链包含1个拷贝的人CD8的所述跨膜域、2个拷贝的人CD28的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。所述第二多肽链包含1个拷贝的人CD8的所述跨膜域、2个拷贝的人4-1BB的所述胞质域和1个拷贝的人CD3ζ的所述胞质域。实例包括由NT25编码的。
令人吃惊且出乎意料的是,(1)如通过流式细胞术测量所测定的,与天然形式的TCR(例如,NT1和NT1b)相比,第二代(例如,NT2、NT3、NT4和NT5)和第三代(例如,NT24)基于抗NY-ESO-1/A2 TCR的CAR在病毒转导的人T细胞上显示出更高的表面表达,(2)与表达天然形式的TCR(例如,NT1、NT1b)的那些相比,表达第二代(例如,NT2、NT3、NT4、NT5)和第三代(例如,NT24)基于抗NY-ESO-1/A2 TCR的CAR的人T细胞与肿瘤靶细胞接合后在TCR-CAR介导的细胞因子分泌(例如,IL-2)和细胞扩增方面更有效,和(3)在涉及异种移植Saos-2肿瘤小鼠模型的体内治疗测定中,数据表明,与天然形式的NT1(NT1a)相比,第二代抗NY-ESO-1TCR-CAR(例如,NT2、NT4)显示出更好的抗肿瘤活性,而第三代抗NY-ESO-1/A2TCR-CAR(例如,NT24)比仅具有来自人CD28(例如,NT2)或仅具有来自4-1BB(例如,NT4)的TCR信号的其第二代对应物部分(例如,NT2、NT4)显著更有效,并且比天然形式的NT1(例如,NT1a)甚至更有效。
在上文所述的抗原受体中,所述抗原结合位点可以以MHC(HLA)限制性方式结合至肿瘤抗原,或肿瘤相关抗原(TAA),或病毒抗原。即,当在细胞上表达时,所述胞外域以MHC(HLA)限制性方式结合至肿瘤抗原,或TAA,或病毒抗原。各种此类抗原是本领域公知的,并且本文列出了一些实例,包括所述肿瘤抗原是NY-ESO-1。
本文公开了制备所述抗原受体和表达其的细胞的方法。所述第一多肽链和第二多肽链可以借助于内部核糖体进入位点(IRES)从两个单独的表达盒或载体或从一个共同的表达盒/载体表达。
两条链可以作为一个融合蛋白表达。因此,本公开内容还提供了一种融合蛋白,其包含所述第一多肽链和所述第二多肽链。在那种情况下,所述第一多肽链和所述第二多肽链可以通过蛋白接头序列或自切割肽序列连接。所述自切割肽序列的实例包括P2A、E2A、F2A或T2A序列。
本公开内容还提供了一种分离的核酸或一组分离的核酸,其编码上文所述的抗原受体或融合蛋白。在一个实施方式中,所述公开内容还提供了一种分离的核酸或一组分离的核酸,其可以包含(I)编码所述第一多肽链的第一核酸序列,所述第一多肽链含有长度至少10个(例如,10、20、30、40、50、60、70、80、100、150、200、250和300个)氨基酸(aa.)的第一多肽区段,和(II)编码所述第二多肽链的第二核酸序列,所述第二多肽链含有长度至少10个(例如,10、20、30、40、50、60、70、80、100、150、200、250和300个)氨基酸的第二多肽区段。所述第一多肽区段与所述第二多肽区段至少90%(例如,95%、96%、97%、98%、99%或100%)相同,和所述第一核酸序列和所述第二核酸序列在密码子内包含至少一个不同的密码子,所述密码子在所述第一多肽区段和所述第二多肽区段中编码相同的氨基酸残基。所述第一多肽链和所述第二多肽链包含长度大于10个氨基酸的相同的多肽序列;和所述第一核酸序列和所述第二核酸序列在所述密码子内包含至少一个不同的密码子,所述密码子编码所述第一核酸序列和所述第二核酸序列的相同多肽序列。在一些实施方式中,至少2%(例如,5%、10%、20%、30%、40%、50%、60%、70%、80%、90%、95%或100%)的所述密码子是不同的。所述第一核酸序列和所述第二核酸序列在所述密码子内小于98%(例如,95%、90%、80%、70%、60%、50%、40%、30%、20%、10%、5%或0%)相同。
在一些实施方式中,所述第一核酸序列和所述第二核酸序列在所述密码子内至少含有一个不同的密码子,所述密码子编码选自以下的一个或多个:(a)所述TCRβ链或其抗原结合片段,(b)所述TCRα链或其抗原结合片段,(c)CD8的所述跨膜域,(d)CD28的所述跨膜域,(e)CD3Z或其片段的所述胞质域,(f)CD3E或其片段的所述胞质域,(g)CD28或其片段的所述胞质域,和(h)4-BB或其片段的所述胞质域。所述分离的核酸可以编码选择以下的一者:SEQ ID NO:9、64、10、11和32-41。所述分离的核酸可以包含选自以下的序列:SEQ IDNO:12、65、13、14和54-63。
上文所述的核酸可以用于表达本文所述的抗原受体。因此,本公开内容还包含一种载体,其包含上文所述的分离的核酸。在一些实施方式中,所述载体是表达载体,其包括如本文所述的病毒载体。当在细胞中表达所述第一多肽链和所述第二多肽链时,可以使用双顺反子或多顺反子表达载体。在用于构建双顺反子或多顺反正载体的各种策略中,IRES已被广泛使用。自切割2A肽也可能是单独使用或与IRES一起使用的良好候选物。因此,所述第一核酸序列和所述第二核酸序列可以通过包含IRES的核酸序列连接。本公开内容还提供了一种细胞,其包含上文所述的抗原受体、融合蛋白、分离的核酸或载体。细胞的实例如下所述,包括淋巴细胞,如T细胞。
还提供了药物组合物,其包含(i)上文所述的核酸、载体或细胞,和(ii)药学上可接受的载体。此类药物组合物可以用于治疗肿瘤或病毒感染疾病的方法中。所述方法包括向有需要的受试者施用有效量的所述药物组合物。
在下文的描述中阐述了本发明的一个或多个实施方式的细节。根据说明和权利要求书,本发明的其他特征、目的和优点将是显而易见的。
附图说明
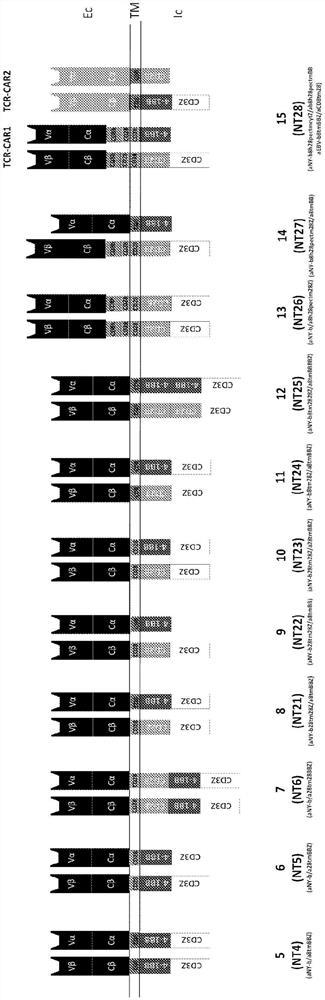
图1A和图1B是显示示例性抗原受体的一组图。(A)四种抗NY-ESO-1TCR或基于TCR的CAR构建体的示意图:1(NT1);2(NT1b);3(NT2);和4(NT3)。(B)另外11种基于抗NY-ESO-1TCR的CAR构建体的示意图:5(NT4);6(NT5);7(NT6);8(NT21);9(NT22);10(NT23);11(NT24);12(NT25);13(NT26);14(NT27);和15(NT28)。
图2A、图2B、图2C和图2D是编码抗NY-ESO-1TCR或基于TCR的CAR构建体的四种核苷酸序列的示意图。A:NT1(SEQ ID NO:12);B:NT1b(SEQ ID NO:65);C:NT2(SEQ ID NO:13);D:NT3(SEQ ID NO:14)。
图3A显示了抗NY-ESO-1TCRα链-2A-TCRβ链(NT1;aNY-TCRa/b)(SEQ ID NO:9)的氨基酸序列。
图3B显示了抗NY-ESO-1TCRβ链-2A-TCRα链(NT1b;aNY-TCRb/a)(SEQ ID NO:64)的氨基酸序列。
图4显示NT2_抗NY-ESO-1βCD28TmCytZCyt-2A-αmuCD28TmCytmuZCyt(aNY-TCR28Z)(SEQ ID NO:10)的氨基酸序列。
图5显示了抗NY-ESO-1βCD28TmCytECyt-2A-αmuCD28TmCytmuECyt(NT3;aNY-TCR28E)(SEQ ID NO:11)的氨基酸序列。
图6显示了核苷酸序列hCD28TmCyt(SEQ ID NO:17)和MuhCD28TmCy(SEQ ID NO:18)的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD28的Tm和Cyt域(CD28TmCyt)(SEQ ID NO:6)。这两条序列之间的总体同源性是56%。相同的核苷酸用星号(*)表示。
图7显示了核苷酸序列hCD3ZCyt(ZCyt)(SEQ ID NO:19)和muhCD3ZCyt(muZCyt)(SEQ ID NO:20)的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD3Z的Cyt域(SEQ ID NO:7)。这两条序列之间的总体同源性是57%。相同的核苷酸用星号(*)表示。
图8显示了核苷酸序列hCD28TmCytCD3ZCyt(28TmCytZCyt)(SEQ ID NO:16)和muhCD28TmCytCD3ZCyt(mu28TmCytZCyt)(SEQ ID NO:21)的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD28的Tm和Cyt域和天然人CD3Z的Cyt(SEQ ID NO:15)。这两条序列之间的总体同源性是59%。相同的核苷酸用星号(*)表示。
图9显示了在人CD3E(Genbank编号:NM_000733.1)(SEQ ID NO:23)的位置119含有单个沉默核苷酸突变(G至C)以破坏BspE 1位点的核苷酸序列hCD3ECyt(ECyt)(SEQ ID NO:23)和muhCD3ECyt(muECyt)(SEQ ID NO:24)的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD3 E链的Cyt域(CD3E)(ECyt)(SEQ ID NO:8)。这两条序列之间的总体同源性是57%。相同的核苷酸用星号(*)表示。
图10显示了CD3Z的Cyt与CD3Z的突变Cyt的核苷酸序列的比对。CD3ZCyt序列(SEQID NO:19)来自人CD3Z(GenBank ID:J04132.1)。hCD3ZCyte和mu2hCD3ZCyt(SEQ ID NO:20)的序列编码相同的氨基酸序列(SEQ ID NO:7)。竖线“|”表示两条序列之间相同的核苷酸。这两条序列之间的总体同源性是60.42%。
图11显示了CD28的Tm与CD28的突变Tm的核苷酸序列的比对。CD28Tm序列(SEQ IDNO:44)来自人CD28(GenBank ID:BC093698.1)。CD28Tm和mu2CD28Tm(SEQ ID NO:45)的序列编码相同的氨基酸序列(SEQ ID NO:27)。竖线“|”表示两条序列之间相同的核苷酸。这两条序列之间的总体同源性是62.96%。
图12显示了CD28的Cyt与CD28的突变Cyt的核苷酸序列的比对。CD28Cyt序列(SEQID NO:46)来自人CD28(GenBank ID:BC093698.1)。hCD28Cyte和mu2hCD28Cyt(SEQ ID NO:47)的序列编码相同的氨基酸序列(SEQ ID NO:28)。竖线“|”表示两条序列之间相同的核苷酸。这两条序列之间的总体同源性是53.65%。
图13显示了4-1BB的Cyt与4-1BB的突变Cyt的核苷酸序列的比对。BBCyt序列(SEQID NO:48)来自人4-1BB(GenBank ID:U03397.1)。hBBCyt和muhBBCyt(SEQ ID NO:49)的序列编码相同的氨基酸序列(SEQ ID NO:29)。竖线“|”表示两条序列之间相同的核苷酸。这两条序列之间的总体同源性是63.49%。
图14显示了hCD8铰链(SEQ ID NO:50)和MuhCD8铰链(SEQ ID NO:51)的核苷酸序列的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD8的铰链域(CD8铰链)(SEQID NO:30)(人CD8A的氨基酸序列135-180(GenBank ID:NM_001768.4))。这两条序列之间的总体同源性是58.70%。使用竖线“|”表示相同的核苷酸。
图15显示了hCD8Tm(SEQ ID NO:52)和MuhCD8Tm(SEQ ID NO:53)的核苷酸序列的比对。这两条DNA序列编码相同的多肽序列,其包含天然人CD8的Tm域(CD8Tm)(SEQ ID NO:31)(人CD8A的氨基酸序列181-206,GenBank ID:NM_001768.4)。这两条序列之间的总体同源性是62.82%。使用竖线“|”表示相同的核苷酸。
图16显示了NT4_抗NY-ESO-1βCD8tmBBCytZCyt-2A-αmuCD8TmBBCytmuZCyt(aNY-TCRBBZ)(SEQ ID NO:32)的序列。
图17显示了NT5_抗NY-ESO-1βCD28tmBBCytZCyt-2A-αmuCD28TmBBCytmuZCyt(aNY-TCRBBZ)(SEQ ID NO:33)的序列。
图18显示了NT6_抗NY-ESO-1βCD28TmCytZCyt-2A-αmuCD28TmCytmuZCyt(aNY-TCR28BBZ)(SEQ ID NO:34)的序列。
图19显示了NT21(SEQ ID NO:35)的氨基酸序列。
图20显示了NT22(SEQ ID NO:36)的氨基酸序列。
图21显示了NT23(SEQ ID NO:37)的氨基酸序列。
图22显示了NT24(SEQ ID NO:38)的氨基酸序列。
图23显示了NT25(SEQ ID NO:39)的氨基酸序列。
图24显示了NT26(SEQ ID NO:40)的氨基酸序列。
图25显示了NT27(SEQ ID NO:41)的氨基酸序列。
图26A和图26B是显示在感染的PG13 VPC上抗NY-ESO-1TCR-CAR的表面表达的图。使用抗NY-ESO-1TCR-CAR+(NT22(载体9;第三代),NT24(载体11;第三代),NT25(载体12;第二代))感染PG13。(A)强度图分别显示了针对FTIC-抗人TCRVb13.1和APC-NYpep/A2四聚体单一或双重阳性细胞的阳性染色。在单个象限中显示了阳性细胞的%。(B)直方图显示了针对NYpep/A2四聚体染色阳性的细胞%。Untd:未感染的。
图27A和图28B是显示在转导的活化人T细胞上天然形式的抗NY-ESO-1TCR或第二代TCR-CAR的表面表达的图。评价的ATC是使用两种抗NY ESO-1TCR(NT1,NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的。(A)强度图分别显示了针对FTIC-抗人TCRVb13.1和APC-NYpep/A2四聚体单一或双重阳性细胞的阳性染色。在单个象限中显示了阳性细胞的百分比。(B)直方图显示了针对NYpep/A2四聚体染色阳性的细胞百分比。
图28是显示使用两种抗NY ESO-1TCR(NT1,NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的ATC对肿瘤细胞(Saos-2)的特异性杀伤作用的一组照片。
图29A和图29B是显示使用两种抗NY ESO-1TCR(NT1,NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的ATC的细胞因子分泌的图。(A)IL-2分泌和(B)INF-γ分泌,其中数据以来自每组中三个样品的平均值±S.D.表示。
图30是显示与肿瘤靶细胞(Saos-2)共培养后使用两种抗NY ESO-1 TCR(NT1,NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的ATC的NYpep/A2+细胞百分比变化的图。
图31A和图31B是显示在转导的活化人T细胞上天然形式的抗NY-ESO-1 TCR或者第二代或第3代TCR-CAR的表面表达的图。评价的ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的,或者未转导的(Untd)。(A)强度图分别显示了针对FITC-抗人TCRVb13.1和APC-NYpep/A2四聚体单一或双重阳性细胞的阳性染色。在单个象限中显示了阳性细胞的%。(B)直方图显示了针对NYpep/A2四聚体染色阳性的细胞%(括号中的数字表示几何平均值)。
图32是显示与靶细胞(Saos-2)接合后使用抗NY ESO-1TCR或四种基于TCR的CAR之一转导的人ATC的靶细胞杀伤作用的图。评价的ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的,或者未转导的(Untd)。
图33A和图33B是显示与靶细胞(Saos-2)接合后使用抗NY ESO-1TCR或四种基于TCR的CAR之一转导的人ATC的(A)IL-2分泌和(B)IFN-γ的图。评价的ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的。ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的,或者未转导的(Untd)。
图34A和图34B是显示与靶细胞(Saos-2)接合后使用抗NY ESO-1TCR或四种基于TCR的CAR之一转导的NYpep/A2+ATC的(A)百分比和(B)扩增变化的图。评价的ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的,或者未转导的(Untd)。
图35A和图35B是显示在异种移植Saos-2肿瘤小鼠模型中在人T细胞中可能增强抗NY-ESO-1/A2 TCR CAR介导的抗肿瘤活性的第二代或第三代TCR-CAR表达的体内治疗作用的图。在体内抗肿瘤治疗实验中评价的ATC是使用抗NY ESO-1TCR(NT1(也称为NT1a);载体1)或抗NY-ESO-1TCR-CAR+(NT2(载体3;第二代),NT4(载体5;第二代),NT5(载体6;第二代),NT24(载体11;第三代))转导的,或者未转导的(Untd)。(A)来自每组中6只小鼠的肿瘤尺寸平均值+S.D.。(B)计算并给出每组小鼠的存活百分比。
具体实施方式
本公开内容涉及赋予和/或增强由细胞免疫疗法(如由过继转移抗原特异性基因修饰的淋巴细胞子集)介导的免疫应答的药剂、方法和组合物。
此类过继细胞转移或过继细胞疗法(ACT)代表了用于治疗癌症患者的有前景的治疗方法。然而,其面临各种障碍,如为了有效的CAR表达和活性而失去稳定性。本公开内容在很多方面解决了这样的障碍,包括但不限于(1)引入例如人4-1BB的共刺激信号传导的第三代TCR-CAR的新颖设计,(2)涉及在不同跨膜域中的变异,相同TCR信号传导元件(如CD3Z以及CD28或4-1BB的共刺激分子)的不同拷贝数的新颖设计。本文所述的方法通过在T细胞中基因表达新型第三代TCR-CAR来增强TCR-CAR介导的T细胞信号传导以改善表达TCR-CAR的T细胞的抗肿瘤能力。
本公开内容提供了包含表达嵌合抗原受体的基因修饰的淋巴细胞的组合物,所述嵌合抗原受体具有调控免疫系统以及先天性和适应性免疫应答的能力。所公开的药剂、方法和组合物提供了具有增强的抗肿瘤功能的基因工程化的淋巴细胞,以及开发此类淋巴细胞的方法。
抗原受体
工程化以表达外来抗原受体的基因修饰的免疫功能细胞(如T细胞和NK细胞)是用于癌症和感染性疾病的有效免疫疗法。分离用于治疗应用的自体抗原特异性免疫细胞(如T细胞)是一项艰巨的任务,并且在此类细胞缺乏或罕见的情况下是不可能的。因此,已经开发了将特异性针对肿瘤或病毒的免疫受体遗传上转移至患者T细胞的策略。为此,已经构建了将抗原(Ag)-识别域连接到TCR或Fc受体的信号传导域的抗原受体。表达这种抗原受体的T细胞再现了由引入的受体介导的免疫特异性应答。
嵌合抗原受体(也称为嵌合免疫受体、嵌合T细胞受体或人工T细胞受体)是已被工程化以使得T细胞具有靶向特定蛋白的新能力的受体蛋白。受体是嵌合的,因为其将抗原结合和T细胞活化功能结合到一个受体中。除了抗原结合位点以外,CAR具有一个或多个功能域。
域
如本文所述,抗原受体包含三个域:胞外域、跨膜域和胞质域(其可以含有胞内信号传导域)。在一些实施方式中,其含有第四个域:在胞外域和胞质域之间的胞外铰链区。这样,嵌合抗原受体将正常T细胞活化的很多方面组合成单一蛋白。其将胞外抗原识别域连接至胞内信号传导域,当抗原结合后,所述胞内信号传导域活化T细胞。
胞外域包含抗原结合或靶点结合域。暴露于细胞外部,该域与潜在靶分子相互作用,并负责将CAR-T细胞靶向表达匹配分子的任何细胞。抗原识别域通常可以来源于作为单链可变片段(scFv)连接在一起的单克隆抗体的可变区或来源于TCR。除了抗体和TCR以外,还可以使用其他方法来引导CAR的特异性,通常利用通常彼此结合的配体/受体对。例如,可以将细胞因子、先天性免疫受体、TNF受体、生长因子和结构蛋白作为抗原识别域。在优选实施方式中,胞外域包含抗体的可变区或其功能性片段,T细胞的胞外域(例如,Va或Vb;VaCa或VbCb)或其功能性片段。
铰链区(也称为间隔区)是位于抗原识别区和细胞外膜之间的小域。理想的铰链区可增强受体的靶点结合域的柔性,从而减少CAR及其靶抗原之间的空间约束。这促进了CAR-T细胞和靶细胞之间的抗原结合和突触形成。铰链序列可以包含来自免疫分子(如IgG、CD8和CD28)的膜近端区域。
跨膜域是一种结构组分,其由跨细胞膜的疏水性α螺旋组成。其将抗原受体锚定在质膜上,将胞外铰链和具有胞质/胞内信号传导区的抗原识别域桥接起来。该域对于整个受体的稳定性是重要的。在本公开内容中,可以使用来自胞质的膜最近端组分的跨膜域,但是不同的跨膜域可以导致不同的受体稳定性。如本文所用,跨膜域可以包含本领域公知的功能性跨膜域(例如,CD3ζ、CD28、CD8、CD4或FcεRiγ的跨膜域,或其变体多肽或功能性片段)。已知CD28跨膜域可导致高度表达的稳定受体。
胞内T细胞信号传导域位于细胞内部的受体胞质域中。在抗原与外部抗原识别域结合后,受体聚集在一起并传输激活信号。然后,受体的内部胞质末端在T细胞内部永久地传递信号。正常的T细胞活化依赖于CD3-ζ胞质域中存在的基于免疫受体酪氨酸活化基序(ITAM)的磷酸化。因此,可以使用CD3-ζ的胞质域。还可以使用其他含有ITAM的域。例如,胞内信号传导域可以包含例如CD3ζ的胞质部分或其功能性片段、FCεRIγ或其功能性片段和/或CD28或其功能性片段。
除了CD3信号传导以外,T细胞还使用共刺激分子,以便在激活后持续存在。因此,CAR受体的胞质域还可以包含一个或多个来自共刺激蛋白的嵌合域(即,共刺激域)。可以使用来自多种共刺激分子的信号传导域。已知通过其结合刺激或增加免疫应答的共刺激多肽的实例包括CD28、OX-40、4-1BB、CD27和NKG2D及其相应配体,包括B7-1、B7-2、OX-40L、4-1BBL、CD70和NKG2D配体。此类多肽存在于肿瘤微环境中并激活对赘生性细胞的免疫应答。在各种实施方式中,通过治疗性转基因促进、刺激或激动促炎性多肽和/或其配体可增强免疫应答细胞的免疫应答。例如,在通过TCR的T细胞活化(信号1)期间的CD28共刺激(信号2)导致持续的增殖、激活诱导的细胞死亡减少(AICD)以及长期淋巴细胞存活改善。下表中列出了可以包含在CAR中的域序列的实例。
表A
用于重新靶向T细胞的两种主要抗原受体是转基因T细胞受体(tTCR)和嵌合抗原受体(CAR)。CAR可以分为两种形式,基于抗体的CAR和基于TCR的CAR。基于抗体的CAR疗法已证明在靶向B细胞白血病方面取得了巨大成功,并且针对实体瘤的试验正在进行中。尽管基于抗体的CAR在癌症免疫疗法中作为治疗剂具有巨大潜力,但其受限于仅识别细胞表面分子的能力。而相反的是,tTCR和基于TCR的CAR具有识别由主要组织相容性复合物(MHC)呈递的任何加工抗原的能力,从而大大扩展了可能的靶标范围。例如,体外研究表明,用内源性出现的NY-ESO-1TCR工程改造的细胞对表达NY-ESO-1的黑色素瘤和非黑色素瘤细胞系具有活性。在最近的一项使用NY-ESO-1导向T细胞的临床试验中,将携带高亲和性tTCR的工程化细胞递送至患有黑色素瘤和滑膜细胞癌的患者。在这项研究中近一半的患者表现出客观临床缓解,凸显了tTCR T细胞在治疗已建立的实体瘤中的潜力。
有两种方式在基因上向T细胞引入与TCR相关的抗原特异性基因:对T细胞进行基因工程化改造以表达外源性天然TCR(例如,TCRα和TCRβ链)和对T细胞进行基因工程化改造以表达基于外源性TCR的CAR。基于TCR的CAR可以通过以下方式建立:其包含胞外域(例如,TCRα链或TCRβ链的胞外域)、跨膜域和TCR信号传导元件(例如,CD3ζ链(CD3Z)或CD3E链(CDE3)的胞质域),其具有或不具有共刺激信号传导元件(如CD28或CD137(4-1BB)的胞质域)的整合。与天然TCR相比,基于TCR的CAR的一个优点是其可以包含TCR信号传导元件,如CD3Z和CDE,并且与共刺激信号传导元件(如CD28和CD137(4-1BB))整合在一起。
已发现在通过TCR(信号1;例如,CD3Z-或CD3E介导的信号传导)的T细胞活化期间,CD28共刺激(信号2)可促进持续的T细胞增殖。为了在单一受体中组合激活和共刺激功能,可以构建由同一分子中的CD3Z和CD28序列组成的CAR。已证明这种IgCD28Z分子在T细胞中具有优异的细胞毒性、增殖以及IL2和IFNγ产生的功能。
基于抗体的CAR的胞外部分(基于抗体的CAR;sFv-CAR)由单链Fv或其片段组成。与sFv-CAR相似,TCR-CAR在特定MHC(HLA)分子的背景下可以包含与TCR或Fc受体的信号传导域连接的TCR可变抗原结合片段(Vα和Vβ,或其抗原结合片段)。TCR-CAR可以以两种基本形式构建:单链TCR-CAR(scTCR-CAR)或双链TCR-CAR(tcTCR-CAR)。对于scTCR-CAR,TCR Vα和Vβ均存在于同一单链TCR蛋白中,而在tcTCR-CAR中,Vα和Vβ处于形成异二聚体的单独的链中。
众所周知,与针对相同同源抗原的抗体相比,TCR对其同源抗原(肽/MHC复合物)的结合亲和性低约100至1,000倍。此外,已报道,与其针对此类抗原的对应母体双链形式的亲和性相比,单链形式的T细胞受体针对其同源抗原通常具有极大降低的亲和性,某些单链T细胞受体完全失去了抗原结合亲和性。关于此类细胞在T细胞活化中的功能,包括诱导增殖、杀死靶细胞和诱导细胞因子分泌,已经证明,包括嵌合TCR在内的TCR的较高亲和性与表达此类受体的经修饰的T细胞的较高效力相关。几份报告表明,单链形式TCR-CAR的结合亲和性太低,以至于没有治疗或诊断价值。因此,为了产生具有相当高的抗原结合亲和性的功能性TCR-CAR,天然TCR的双链形式比单链形式具有更显著的优势。
核酸之间的重组是分子生物学中公认的现象。通常将需要参与的核酸序列之间具有较强序列同源性的遗传重组称为同源重组。尽管大多数基因敲除策略都采用同源重组来实现有针对性的敲除,但在某些系统中,基因重组的发生会不利地影响基因操作。特别地,同源重组事件可不利地影响载体,特别是病毒载体(例如,腺病毒、逆转录病毒、腺相关病毒、疱疹病毒等)的构建和产生,其中在例如病毒载体通过一个或多个宿主细胞和/生物体传代和/或增殖期间,在单个、稳定病毒载体内保持高度同源的序列(例如,相同多肽序列)而没有同源重组通常是理想的。
作为减少可能的病毒重组事件的方式,可以采用双载体方法将包含由相似的高度同源的核酸序列编码的高度同源的多肽序列的两个蛋白分子转导至单个细胞。例如,双链形式的TCR-CAR,其分别由各种VaCa和VbCb组成,但是共享相同的信号传导元件多肽序列(例如,CD28和/或CD3Z的胞质域)。两种不同载体,各自编码两个目标同源蛋白之一,可以从单独VPC中产生。然而,单一载体向哺乳动物细胞(如暴露于逆转录病毒的活化的T细胞)的成功转导率通常是有限的。
克服与低宿主细胞转导效率有关的此类问题的新方法是使得能够在单个病毒载体上递送两个或更多个编码高度同源(例如,相同)多肽的核酸序列。这种方法允许人们生产包含编码两种或更多种高度同源(例如,相同)多肽或其多肽域的核酸序列的病毒载体序列。即使在例如在宿主细胞中延长传代和/或多次感染、染色体整合和/或切除事件期间,所述病毒载体序列也具有降低的诸如核酸序列之间的同源重组的风险。此类降低的同源重组率至少部分归因于如在本文中所述的病毒载体合成期间对遗传密码简并性的开发。
此外,铰链/间隔区由CAR的非抗原结合胞外区组成。铰链/间隔区通过提供柔性、延长长度、允许发生二聚化或提高稳定性来调节CAR功能。这些性质已表明影响效应细胞与靶细胞的相互作用,从而影响激活信号的强度。常规铰链/间隔区使用免疫球蛋白Fc、CD8α和CD28间隔区制备。
下文列出了可以包括在本文公开的CAR中的域序列的实例和相关的核酸序列。
SEQ ID NO:1:1G4 a95LY TCR的α链(275aa)
METLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSCDVKLVEKSFETDTNLNFQNLSVIGFRILLLKVAGFNLLMTLRLWSSR
SEQ ID NO:2:1G4 a95LY TCR的β链(311aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADCGFTSESYQQGVLSATILYEILLGKATLYAVLVSALVLMAMVKRKDSRG
SEQ ID NO:3:P2A(19aa)
ATNFSLLKQAGDVEENPGP
SEQ ID NO:4:1G4 a95LY TCR的α链的Ec域(227aa)
METLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESS
SEQ ID NO:5:1G4 a95LY TCR的β链的Ec域(262aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRAD
SEQ ID NO:6:人CD28TmCyt(68aa)
FWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS
SEQ ID NO:7:人CD3ZCyt(112aa)
RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:8:人CD3ECyt(55aa)
KNRKAKAKPVTRGAGAGGRQRGQNKERPPPVPNPDYEPIRKGQRDLYSGLNQRRI
SEQ ID NO:9:NT1_aNY-TCR(612aa)
METLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSCDVKLVEKSFETDTNLNFQNLSVIGFRILLLKVAGFNLLMTLRLWSSRAKRSGSGATNFSLLKQAGDVEENPGPMSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADCGFTSESYQQGVLSATILYEILLGKATLYAVLVSALVLMAMVKRKDSRG
SEQ ID NO:64:NT1b_Xho_NY-ESO.txt.xprt(612aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADCGFTSESYQQGVLSATILYEILLGKATLYAVLVSALVLMAMVKRKDSRGAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSCDVKLVEKSFETDTNLNFQNLSVIGFRILLLKVAGFNLLMTLRLWSSR
SEQ ID NO:10:NT2_aNY-TCR28Z(NYESO1-TCRba28Z_Xho-NY-ESO1
bmu2CD28tmcytCD3Zcyt-P2A-1g4TCRa95LYCD28tmcytCD3Zcyt),883aa
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:11:NT3_aNY-TCR28E
(NYESO1-TCRba28E_NY-ESO1bmu2CD28tmcytCD3Ecyt-P2A-1g4TCRa95LYCD28tmcytCD3Ecyt)(769aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSKNRKAKAKPVTRGAGAGGRQRGQNKERPPPVPNPDYEPIRKGQRDLYSGLNQRRIAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSKNRKAKAKPVTRGAGAGGRQRGQNKERPPPVPNPDYEPIRKGQRDLYSGLNQRRI
SEQ ID NO:12:编码NT1_aNY-TCR
(NYESO1-TCRab_Xho-NY-ESO11g4TCRa95LY-P2A-b-Not)的核苷酸序列
ATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCTGTGATGTCAAGCTGGTCGAGAAAAGCTTTGAAACAGATACGAACCTAAACTTTCAAAACCTGTCAGTGATTGGGTTCCGAATCCTCCTCCTGAAAGTGGCCGGGTTTAATCTGCTCATGACGCTGCGGCTGTGGTCCAGCCGGGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACTGTGGCTTCACCTCCGAGTCTTACCAGCAAGGGGTCCTGTCTGCCACCATCCTCTATGAGATCTTGCTAGGGAAGGCCACCTTGTATGCCGTGCTGGTCAGTGCCCTCGTGCTGATGGCTATGGTCAAGAGAAAGGATTCCAGAGGCTAA
SEQ ID NO:65:NT1b_Xho_NY-ESO1-TCRb-P2A-1g4TCRa95LY-not.txt.xdna
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACTGTGGCTTCACCTCCGAGTCTTACCAGCAAGGGGTCCTGTCTGCCACCATCCTCTATGAGATCTTGCTAGGGAAGGCCACCTTGTATGCCGTGCTGGTCAGTGCCCTCGTGCTGATGGCTATGGTCAAGAGAAAGGATTCCAGAGGCGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCTGTGATGTCAAGCTGGTCGAGAAAAGCTTTGAAACAGATACGAACCTAAACTTTCAAAACCTGTCAGTGATTGGGTTCCGAATCCTCCTCCTGAAAGTGGCCGGGTTTAATCTGCTCATGACGCTGCGGCTGTGGTCCAGCCGGTAA
SEQ ID NO:13:编码NT2_aNY-TCR28Z NYESO1-TCRba28Z_Xho-NY-ESO1bmu2CD28tmcytCD3Zcyt-P2A-1g4TCRa95LYCD28tmcytCD3Zcyt-Not的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:14:编码NT3_aNY-TCR28E
NYESO1-TCRba28E_Xho-NY-ESO1
bmu2CD28tmcytCD3Ecyt-P2A-1g4TCRa95LYCD28tmcytCD3Ecyt-Not的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTAAAAACCGCAAAGCTAAAGCTAAACCCGTCACTAGGGGGGCCGGAGCAGGAGGGCGCCAGCGCGGTCAGAATAAAGAACGCCCTCCTCCCGTCCCTAATCCTGATTACGAACCGATTAGAAAGGGGCAAAGAGATCTCTACAGCGGACTCAACCAACGGAGAATTGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCAAGAATAGAAAGGCCAAGGCCAAGCCTGTGACACGAGGAGCGGGTGCTGGCGGCAGGCAAAGGGGACAAAACAAGGAGAGGCCACCACCTGTTCCCAACCCAGACTATGAGCCCATCCGCAAAGGCCAGCGGGACCTGTATTCTGGCCTGAATCAGAGACGCATCTAA
SEQ ID NO:15:hCD28TmCytCD3ZCyt(141aa)
MNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:16:编码hCD28TmCytCD3ZCyt的核苷酸序列(540bp)
TTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGC
SEQ ID NO:17:编码hCD28TmCyt的核苷酸序列(204bp)
TTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCC
SEQ ID NO:18:编码hCD28TmCytCD3ZCyt的突变的核苷酸序列(mu28TmCyt)(204bp)
TTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGT
SEQ ID NO:19:编码人CD3ZCyt(ZCyt)的核苷酸序列336bp
AGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGC
SEQ ID NO:20:编码人CD3ZCyt的突变的核苷酸序列(muZCyt),muhCD3ZCyt,336bp
CGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGA
SEQ ID NO:21:编码hCD28TmCytCD3ZCyt(mu28TmCytmuCyt)muhCD28TmCytmuCD3ZCyt的突变的核苷酸序列,540bp
TTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGA
SEQ ID NO:22:人CD3ECyt_NM_000733.1的核苷酸序列(165bp)
AAGAATAGAAAGGCCAAGGCCAAGCCTGTGACACGAGGAGCGGGTGCTGGCGGCAGGCAAAGGGGACAAAACAAGGAGAGGCCACCACCTGTTCCCAACCCAGACTATGAGCCCATCCGGAAAGGCCAGCGGGACCTGTATTCTGGCCTGAATCAGAGACGCATC
SEQ ID NO:23:人CD3ECyt_NM_000733.1的核苷酸序列,在位置119具有单一沉默核苷酸突变(G至C),以破坏BspE 1位置(165bp)
AAGAATAGAAAGGCCAAGGCCAAGCCTGTGACACGAGGAGCGGGTGCTGGCGGCAGGCAAAGGGGACAAAACAAGGAGAGGCCACCACCTGTTCCCAACCCAGACTATGAGCCCATCCGCAAAGGCCAGCGGGACCTGTATTCTGGCCTGAATCAGAGACGCATC
SEQ ID NO:24:沉默突变的人CD3ECyt_NM_000733.1的核苷酸序列,已编辑,165bp
AAAAACCGCAAAGCTAAAGCTAAACCCGTCACTAGGGGGGCCGGAGCAGGAGGGCGCCAGCGCGGTCAGAATAAAGAACGCCCTCCTCCCGTCCCTAATCCTGATTACGAACCGATTAGAAAGGGGCAAAGAGATCTCTACAGCGGACTCAACCAACGGAGAATT
SEQ ID NO:25:对应于人NY-ESO-1的残基157至165的人NY-ESO-1肽(NY-ESO-1:157-165)(9aa)
SLLMWITQC
SEQ ID NO:26:CD28pec(40aa)
KIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKP
SEQ ID NO:27:人CD28 Tm
FWVLVVVGGVLACYSLLVTVAFIIFWV
SEQ ID NO:28:人CD28 Cyt
RSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS
SEQ ID NO:29:人4-1BBCyt
KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL
SEQ ID NO:30:CD8铰链.txt.xprt(46aa)
AKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFA
SEQ ID NO:31:CD8Tm_aa 181-20.txt.xprt(26aa)
CDIYIWAPLAGTCGVLLLSLVITLYC
SEQ ID NO:32:NT4_NYESO1-TCRbaBBZ_Xho-NY-ESO1 bmuCD8tmBBcyt CD3Zcyt-P2A-1g4TCRa95LYCCD8tmBBcytZcyt-Not.txt.xprt(883aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:33:NT5_设计_NYE.txt.xprt(885aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:34:NT6_NYESO1-TCRb.txt.xprt(967 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:35:NT21_.txt.xprt(883 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:36:NT22_.txt.xprt(771 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL
SEQ ID NO:37:NT23_.txt.xprt(884 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKFWVLVVVGGVLACYSLLVTVAFIIFWVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:38:NT24_.txt.xprt(882 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKCDIYIWAPLAGTCGVLLLSLVITLYCRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:39:NT25_.txt.xprt(965 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKCDIYIWAPLAGTCGVLLLSLVITLYCRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:40:NT26_.txt.xprt(1059 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFAPRKIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKPFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFAPRKIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKPFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
SEQ ID NO:41:NT27_.txt.xprt(859 aa)
MSIGLLCCAALSLLWAGPVNAGVTQTPKFQVLKTGQSMTLQCAQDMNHEYMSWYRQDPGMGLRLIHYSVGAGITDQGEVPNGYNVSRSTTEDFPLRLLSAAPSQTSVYFCASSYVGNTGELFFGEGSRLTVLEDLKNVFPPEVAVFEPSEAEISHTQKATLVCLATGFYPDHVELSWWVNGKEVHSGVSTDPQPLKEQPALNDSRYALSSRLRVSATFWQDPRNHFRCQVQFYGLSENDEWTQDRAKPVTQIVSAEAWGRADGSPKAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFAPRKIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKPFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRAKRSGSGATNFSLLKQAGDVEENPGPMETLLGLLILWLQLQWVSSKQEVTQIPAALSVPEGENLVLNCSFTDSAIYNLQWFRQDPGKGLTSLLLIQSSQREQTSGRLNASLDKSSGRSTLYIAASQPGDSATYLCAVRPLYGGSYIPTFGRGTSLIVHPYIQNPDPAVYQLRDSKSSDKSVCLFTDFDSQTNVSQSKDSDVYITDKTVLDMRSMDFKSNSAVAWSNKSDFACANAFNNSIIPEDTFFPSPESSGSPKCDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL
SEQ ID NO:42:hCD28_人CD28_完整cds_mRNA_BC093698.1.txt.xdna的pEc的核苷酸序列(120bp)
AAAATTGAAGTTATGTATCCTCCTCCTTACCTAGACAATGAGAAGAGCAATGGAACCATTATCCATGTGAAAGGGAAACACCTTTGTCCAAGTCCCCTATTTCCCGGACCTTCTAAGCCC
SEQ ID NO:43:mu2 hCD28pEc_人CD28_完整cds_mRNA_BC093698.1.txt.xdna的nt(120bp)
AAGATCGAGGTAATGTACCCACCGCCCTATCTTGATAACGAAAAATCTAACGGTACAATAATTCACGTCAAGGGCAAGCATTTGTGCCCTTCCCCGTTGTTCCCGGGCCCAAGCAAACCG
SEQ ID NO:44:CD28Tm_人CD28_BC093698.1.txt.xdna的核苷酸序列(81bp)
TTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTG
SEQ ID NO:45:mu2CD28Tm_人CD28_BC093698.1.txt.xdna的核苷酸序列(81bp)
TTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTC
SEQ ID NO:46:hCD28Cyt.txt.xdna的核苷酸序列(123bp)
AGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCC
SEQ ID NO:47:mu2hCD28Cyt.txt.xdna的核苷酸序列(123bp)
CGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGT
SEQ ID NO:48:设计_h4-1BBCyt.xdna的核苷酸序列(126bp)
AAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTG
SEQ ID NO:49:设计_muh41BBCyt.xdna的核苷酸序列(126bp)
AAGAGAGGGCGTAAAAAGCTGCTCTACATCTTTAAGCAGCCTTTCATGCGTCCTGTTCAGACAACACAGGAAGAGGACGGATGCTCTTGCAGGTTCCCTGAGGAGGAGGAGGGTGGTTGCGAGCTC
SEQ ID NO:50:CD8铰链的核苷酸序列(138bp)
GCTAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCC
SEQ ID NO:51:muCD8铰链的核苷酸序列(138bp)
GCTAAGCCCACTACTACCCCAGCTCCCAGGCCTCCCACACCTGCCCCAACAATCGCCAGCCAGCCACTGTCCCTTAGGCCCGAGGCCTGTAGGCCCGCCGCCGGAGGAGCCGTGCACACCCGCGGACTGGATTTTGCT
SEQ ID NO:52:H_CD8A_NM_001768.4.xdna的CD8Tm_aa 181-206的核苷酸序列(78bp)
TGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGC
SEQ ID NO:53:MuCD8Tm的核苷酸序列,78bp
MuCD8Tm_aa 181-206of H_CD8A_NM_001768.4
TGCGACATTTATATTTGGGCCCCTCTCGCTGGCACATGCGGCGTGTTGTTGCTCAGCCTCGTGATTACACTTTATTGT
SEQ ID NO:54:NT4_设计_NYESO1-TCRbaBBZ_Xho-NY-ESO1
bmuCD8tmBBcytCD3Zcyt-P2A-1g4TCRa95LYCCD8tmBBcytZcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTGCGACATTTATATTTGGGCCCCTCTCGCTGGCACATGCGGCGTGTTGTTGCTCAGCCTCGTGATTACACTTTATTGTAAGAGAGGGCGTAAAAAGCTGCTCTACATCTTTAAGCAGCCTTTCATGCGTCCTGTTCAGACAACACAGGAAGAGGACGGATGCTCTTGCAGGTTCCCTGAGGAGGAGGAGGGTGGTTGCGAGCTCCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:55:NT5_设计_NYESO1-TCRbaCD28tmBBZ_Xho-NY-ESO1
bmu2CD28tmBBcytCD3Zcyt-P2A-1g4TCRa95LYCD28tmBBcytCD3Zcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCAAGAGAGGGCGTAAAAAGCTGCTCTACATCTTTAAGCAGCCTTTCATGCGTCCTGTTCAGACAACACAGGAAGAGGACGGATGCTCTTGCAGGTTCCCTGAGGAGGAGGAGGGTGGTTGCGAGCTCCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:56:NT6_NYESO1-TCRba28BBZ_Xho-NY-ESO1
bmu2CD28tmcytBBcytCD3Zcyt-P2A-1g4TCRa95LYCD28tmcytBBcytCD3Zcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTAAGAGAGGGCGTAAAAAGCTGCTCTACATCTTTAAGCAGCCTTTCATGCGTCCTGTTCAGACAACACAGGAAGAGGACGGATGCTCTTGCAGGTTCCCTGAGGAGGAGGAGGGTGGTTGCGAGCTCCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:57:NT21_NT2-4h1_NYESO1-TCRb28tmcytZa8tmBZh1_Xho-NY-ESO1bmu2CD28tmcytCD3Zcyt-P2A-1g4TCRa95LYCD8tmBBcytCD3Zcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:58:NT22_NT2-4h2_NYESO1-TCRb28tmcytZa8tmBh2_Xho-NY-ESO1bmu2CD28tmcytCD3Zcyt-P2A-1g4TCRa95LYCD8tmBBcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGTAA
SEQ ID NO:59:NT23_NT2-5h1_NYESO1-TCRb28tmcytZa28tmBZh1_Xho-NY-ESO1bmu2CD28tmcytCD3Zcyt-P2A-1g4TCRa95LYCD28tmBBcytCD3Zcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:60:NT24_NT2-4h3_aNY-TCR-CAR b8tm28cytZa8tmBBcytZ_Xho-NY-ESO1bmuCD8tmmu2CD28cytmuCD3Zcyt-接头
P2A-1g4TCRa95LYCD8tmBBcytZcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTGCGACATTTATATTTGGGCCCCTCTCGCTGGCACATGCGGCGTGTTGTTGCTCAGCCTCGTGATTACACTTTATTGTCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:61:NT25_aNY-TCR-CAR b8tm28cytcyt-a8tmBBcytcytZ_Xho-NY-ESO1bmu2CD8tmCD28cytmu2CD28cytmu2CD3Zcyt-接头
P2A-1g4TCRa95LYCD8tmmuBBcytBBcytZcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGTGCGACATTTATATTTGGGCCCCTCTCGCTGGCACATGCGGCGTGTTGTTGCTCAGCCTCGTGATTACACTTTATTGTAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAGAGAGGGCGTAAAAAGCTGCTCTACATCTTTAAGCAGCCTTTCATGCGTCCTGTTCAGACAACACAGGAAGAGGACGGATGCTCTTGCAGGTTCCCTGAGGAGGAGGAGGGTGGTTGCGAGCTCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:62:NT26_aNY-TCR-CARb8h28pectmcytZa8h28pectmcytZ_Xho-NY-ESO1bmu2CD8hmu2CD28pectmcytmu2Zcyt-P2A-1g4TCRa95LYCD8hCD28pectmcytZcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGGCAAAACCGACGACCACCCCTGCCCCCAGGCCTCCTACTCCCGCCCCGACGATTGCCAGCCAACCGTTAAGTTTAAGACCGGAAGCATGTAGACCGGCAGCTGGTGGGGCTGTTCATACACGTGGCTTAGATTTTGCGCCTAGGAAGATCGAGGTAATGTACCCACCGCCCTATCTTGATAACGAAAAATCTAACGGTACAATAATTCACGTCAAGGGCAAGCATTTGTGCCCTTCCCCGTTGTTCCCGGGCCCAAGCAAACCGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAAGCTAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCCCTAGGAAAATTGAAGTTATGTATCCTCCTCCTTACCTAGACAATGAGAAGAGCAATGGAACCATTATCCATGTGAAAGGGAAACACCTTTGTCCAAGTCCCCTATTTCCCGGACCTTCTAAGCCCTTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCTAA
SEQ ID NO:63:NT27_NT2b-4h1_aNY-TCR-CAR
bCD8h28pectmcytZa8tmBBcyt_Xho-NY-ESO1bmu2CD8hmu2CD28pectmcytmu2Zcyt-P2A-1g4TCRa95LYCD8tmBBcyt-Not.xdna的核苷酸序列
ATGAGCATCGGCCTCCTGTGCTGTGCAGCCTTGTCTCTCCTGTGGGCAGGTCCAGTGAATGCTGGTGTCACTCAGACCCCAAAATTCCAGGTCCTGAAGACAGGACAGAGCATGACACTGCAGTGTGCCCAGGATATGAACCATGAATACATGTCCTGGTATCGACAAGACCCAGGCATGGGGCTGAGGCTGATTCATTACTCAGTTGGTGCTGGTATCACTGACCAAGGAGAAGTCCCCAATGGCTACAATGTCTCCAGATCAACCACAGAGGATTTCCCGCTCAGGCTGCTGTCGGCTGCTCCCTCCCAGACATCTGTGTACTTCTGTGCCAGCAGTTACGTCGGGAACACCGGGGAGCTGTTTTTTGGAGAAGGCTCTAGGCTGACCGTACTGGAGGACCTGAAAAACGTGTTCCCACCCGAGGTCGCTGTGTTTGAGCCATCAGAAGCAGAGATCTCCCACACCCAAAAGGCCACACTGGTGTGCCTGGCCACAGGCTTCTACCCCGACCACGTGGAGCTGAGCTGGTGGGTGAATGGGAAGGAGGTGCACAGTGGGGTCAGCACAGACCCGCAGCCCCTCAAGGAGCAGCCCGCCCTCAATGACTCCAGATACGCTCTGAGCAGCCGCCTGAGGGTCTCGGCCACCTTCTGGCAGGACCCCCGCAACCACTTCCGCTGTCAAGTCCAGTTCTACGGGCTCTCGGAGAATGACGAGTGGACCCAGGATAGGGCCAAACCCGTCACCCAGATCGTCAGCGCCGAGGCCTGGGGTAGAGCAGACGGCTCTCCTAAGGCAAAACCGACGACCACCCCTGCCCCCAGGCCTCCTACTCCCGCCCCGACGATTGCCAGCCAACCGTTAAGTTTAAGACCGGAAGCATGTAGACCGGCAGCTGGTGGGGCTGTTCATACACGTGGCTTAGATTTTGCGCCTAGGAAGATCGAGGTAATGTACCCACCGCCCTATCTTGATAACGAAAAATCTAACGGTACAATAATTCACGTCAAGGGCAAGCATTTGTGCCCTTCCCCGTTGTTCCCGGGCCCAAGCAAACCGTTCTGGGTTCTCGTCGTCGTGGGAGGTGTGTTAGCATGTTACTCTCTCTTGGTTACTGTCGCTTTCATAATCTTTTGGGTCCGCTCAAAACGCTCTCGCTTGTTACATTCCGATTATATGAATATGACACCTAGGAGACCTGGCCCGACTAGGAAACACTATCAACCTTACGCACCTCCCAGAGATTTTGCTGCTTACAGGAGTCGGGTCAAATTTTCACGCTCCGCTGATGCTCCTGCCTATCAACAAGGGCAAAATCAATTGTACAATGAATTGAACTTGGGTAGAAGGGAAGAATATGACGTGCTCGATAAACGGAGGGGGAGAGATCCAGAAATGGGCGGTAAACCACGGCGCAAAAATCCACAAGAGGGATTGTATAACGAGCTCCAAAAGGACAAAATGGCAGAAGCTTATTCAGAAATAGGAATGAAGGGGGAAAGGAGACGAGGTAAAGGTCATGACGGATTGTATCAAGGATTGTCAACCGCTACTAAAGATACATATGATGCTTTGCATATGCAAGCTTTGCCTCCCAGAGCCAAGCGGTCTGGGTCTGGGGCCACCAACTTCAGCCTGCTGAAGCAGGCCGGCGACGTGGAGGAGAACCCCGGCCCCATGGAGACCCTCTTGGGCCTGCTTATCCTTTGGCTGCAGCTGCAATGGGTGAGCAGCAAACAGGAGGTGACGCAGATTCCTGCAGCTCTGAGTGTCCCAGAAGGAGAAAACTTGGTTCTCAACTGCAGTTTCACTGATAGCGCTATTTACAACCTCCAGTGGTTTAGGCAGGACCCTGGGAAAGGTCTCACATCTCTGTTGCTTATTCAGTCAAGTCAGAGAGAGCAAACAAGTGGAAGACTTAATGCCTCGCTGGATAAATCATCAGGACGTAGTACTTTATACATTGCAGCTTCTCAGCCTGGTGACTCAGCCACCTACCTCTGTGCTGTGAGGCCCCTGTACGGAGGAAGCTACATACCTACATTTGGAAGAGGAACCAGCCTTATTGTTCATCCGTATATCCAGAACCCTGACCCTGCCGTGTACCAGCTGAGAGACTCTAAATCCAGTGACAAGTCTGTCTGCCTATTCACCGATTTTGATTCTCAAACAAATGTGTCACAAAGTAAGGATTCTGATGTGTATATCACAGACAAAACTGTGCTAGACATGAGGTCTATGGACTTCAAGAGCAACAGTGCTGTGGCCTGGAGCAACAAATCTGACTTTGCATGTGCAAACGCCTTCAACAACAGCATTATTCCAGAAGACACCTTCTTCCCCAGCCCAGAAAGTTCCGGCTCCCCAAAATGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAAACGGGGCAGAAAGAAACTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGTAA
组合物和试剂盒
在一个方面中,本公开内容提供了一种组合物,其包含多个如上文所述的表达嵌合抗原受体或其链的基因修饰的淋巴细胞,用于调节受试者的免疫系统。
在本发明中可以使用各种淋巴细胞。淋巴细胞的实例可以包括T细胞、B细胞、NK细胞、巨噬细胞、中性粒细胞、树突状细胞、肥大细胞、嗜酸性粒细胞和嗜碱性粒细胞。在一些实施方式中,淋巴细胞来自CD34造血干细胞、胚胎干细胞或诱导型多能干细胞。淋巴细胞可以是自体的、同种异体的、同基因的或异种的。在一些实施方式中,淋巴细胞是自体的。在一些实施方式中,淋巴细胞是人淋巴细胞。
在一些实施方式中,淋巴细胞是外周血淋巴细胞(PBL)。在一些实施方式中,淋巴细胞可以是肿瘤浸润淋巴细胞(TIL)。在一些实施方式中,淋巴细胞可以表达嵌合抗原受体。在一些实施方式中,淋巴细胞可以表达重组T细胞受体。CAR或TCR可以结合至目标癌抗原。
此类癌和/或肿瘤抗原的实例包括但不限于,例如,细胞周期蛋白依赖性激酶-4、β-连环蛋白、Caspase-8、MAGE-1、MAGE-3、酪氨酸酶、表面Ig独特型、Her-2/neu、MUC-1、HPVE6、HPV E7、CD5、独特型、CAMPATH-1、CD20、CEA、粘蛋白-1、Lewisx、CA-125、EGFR、p185HER2、IL-2R、FAP,、腱生蛋白、金属蛋白酶、phCG、gp100或Pmell7、HER2/neu、CEA、gp100、MART1、TRP-2、黑色素-A、NY-ESO-1、MN(gp250)、独特型、MAGE-1、MAGE-3、酪氨酸酶、端粒酶、MUC-1抗原和生殖细胞来源的抗原、血型抗原,例如,Lea、Leb、LeX、LeY、H-2、B-1、B-2抗原。
在某些实施方式中,表达相同CAR的T细胞可以结合一种以上的癌症和/或肿瘤抗原;例如,T细胞的一种CAR与MAGE抗原的结合可以与T细胞的另一种CAR抗原(如黑色素A、酪氨酸酶或gp100)的结合组合。例如,CD20是在恶性和非恶性B细胞表面上均存在的泛B抗原,已证实其是治疗非霍奇金氏淋巴瘤的免疫治疗抗体的极为有效的靶点。在这一方面,在本发明的意义内,泛T细胞抗原(如CD2、CD3、CD5、CD6和CD7)也包含肿瘤相关抗原。其他示例性肿瘤相关抗原包括但不限于MAGE-1、MAGE-3、MUC-1、HPV 16、HPV E6&E7、TAG-72、CEA、L6-抗原、CD19、CD22、CD37、CD52、HLA-DR、EGF受体和HER2受体。在很多情况下,在文献中已经报道了针对这些抗原的每一种的免疫反应性抗体(和/或免疫反应性抗原结合片段)。
在实施方式中,嵌合抗原受体的两条链由一个核酸转基因编码。两条链可以通过自切割肽序列连接。或者,编码两条链的两条序列通过包含IRES的核酸序列连接,以使得两条链可以分别翻译。转基因的表达可以由组成型激活的启动子或由诱导型启动子调控。在一些实施方式中,转基因的表达可以由淋巴细胞的活化状态诱导。在其他情况下,可以通过具有整合能力的γ逆转录病毒或慢病毒、DNA转座等将转基因导入淋巴细胞。
可以将上文所述的基因修饰的淋巴细胞引入适于施用的药物组合物。药物组合物通常包含基本上分离的/纯的淋巴细胞和以适于向受试者施用形式的药学上可接受的载体。药学上可接受的载体可以部分地通过所施用的特定组合物以及用于施用所述组合物的特定方法来确定。药物组合物的配制通常完全符合美国食品和药物管理局的药品生产质量管理规范(GMP)的规定。
涉及组合物、载体、稀释剂和试剂的术语“药学上可接受的”可以互换使用,并且包括能够向受试者施用或者向受试者施用后不会产生其程度将会阻止组合物的施用的不希望的生理作用的物质。例如,“药学上可接受的赋形剂”包括在制备药物组合物中使用的通常是安全、无毒和所需的赋形剂,并且包括兽用以及人用药使用的可接受的赋形剂。
此类载体或稀释剂的实例包括但不限于水、盐水、林格氏溶液、右旋糖溶液和5%人血清白蛋白。用于药物活性物质的此类介质和化合物的用途是本领域熟知的。除非任何常规介质或化合物与所公开的组合物不相容,否则考虑将其用于组合物中。在一些实施方式中,也可以将第二治疗剂(如抗癌或抗肿瘤)加入药物组合物中。
适于注射用的药物组合物包括无菌水溶液(水溶性)或分散液,以及用于临时制备无菌注射溶液或分散液的无菌粉末。对于静脉内施用,适宜载体包括生理盐水、抑菌水、Cremophor ELTM(BASF,Parsippany,N.J.)或磷酸盐缓冲盐水(PBS)。在所有情况下,组合物必须是无菌的,并且应该以易于注射的程度存在的流体。其必须在生产和储存条件下保持稳定,并且必须进行防腐处理以防止微生物(如细菌和真菌)的污染。载体可以是溶剂或分散介质,其包括,例如,水、乙醇、多元醇(例如,甘油、丙二醇和液体聚乙二醇等)及其适宜的混合物。可以例如通过使用诸如卵磷脂的包衣,在分散液的情况下通过维持所需的粒径以及通过使用表面活性剂来维持适当的流动性。在一些实施方式中,组合物包含如上文所述的基因修饰的淋巴细胞和任选地冻干保护剂(例如,甘油、DMSO、PEG)。
可以在试剂盒中提供本文所述的组合物或药物组合物。在一个实施方式中,试剂盒包括(a)含有组合物的容器和任选地(b)信息材料。信息材料可以是与本文所述的方法和/或药剂用于治疗获益的用途有关的描述性、说明性、市场营销或其他材料。例如,试剂盒可以包括针对生产,针对所使用的治疗方案和给药期的说明。在一个实施方式中,试剂盒还可以包括另外的治疗剂(例如,检查点调节剂)。试剂盒可以包含一个或多个容器,每个含有不同药剂。例如,试剂盒包含含有组合物的第一容器和含有另外的治疗剂的第二容器。
容器可以包含单位剂量的药物组合物。除了组合物以外,试剂盒还包含其他成分,如溶剂或缓冲剂、佐剂、稳定剂或防腐剂。试剂盒任选地包含适于施用组合物的装置,例如,注射器或其他适宜的递送装置。所述装置可以预先装有一种或两种药剂,或者可以是空的,但适于装载。
用于制备组合物的方法
在通常情况下,除非另有说明,否则本发明的实施采用化学、分子生物学、重组DNA技术、PCR技术、免疫学(例如,抗体技术)、表达系统(例如,无细胞表达、噬菌体展示、核糖体展示和PROFUSION)的常规技术,以及在本领域技术范围内并在文献中进行了说明的任何必要的细胞培养。尽管本发明的某些方面涉及重组RNA逆转录病毒(例如,慢病毒HIV-2、SIV等)的组合物和用途,但是可以使用前病毒(proviral)DNA克隆进行分子克隆,从而允许使用标准克隆技术。可以根据本领域公知的方法进行通过合成寡聚脱氧核苷酸在体外的定点诱变。可以通过本领域公知的方法,例如,通常依赖于在PCR扩增期间使用融合引物(其任选地是致突变的)的基因SOE(通过重叠延伸剪接)方法(Horton等,1989Gene 77:61-68;美国专利号5,023,171)制备本发明的基因融合(特别是在融合蛋白的合成中使用的,例如CAR)。可以根据生产厂商的说明书使用DNA热循环仪通过PCR技术进行DNA片段的酶促扩增。
可以通过测序来进行核苷酸序列的验证。可以通过任何本领域公知的方法,来验证是否已经在本发明的单个载体中包含的两个同源多肽之间发生了同源重组事件,包括但不限于Northern印迹和/或RT-PCR方法(例如,如果直接在分离的逆转录基因组内进行评估)、Southern印迹和/或PCR方法(例如,对包含整合的逆转录病毒载体的宿主细胞基因组DNA进行评估)和SDS-PAGE随后进行Western印迹和/或免疫沉淀随后进行SDS-PAGE并检测标记的多肽(例如,如果同源多肽具有可辨别的尺寸和/或包含可区分的域、特征和/或表位)
一些实施方式涉及建立编码带有简并密码子的两个或更多个相同或高度同源分子的逆转录病毒载体。此前已经在例如美国专利号9,206,440和Im EJ等,Recombination–deletion between homologous cassettes in retrovirus is suppressed via astrategy of degenerate codon substitution.Molecular Therapy-Methods&ClinicalDevelopment(2014)文章编号:14022中描述了使用沉默突变方法来建立编码带有简并密码子的两个或更多个相同或高度同源分子的逆转录病毒载体的方法以减少可能的DNA重组事件。参考文献通过引用并入。
在另一个方面中,本公开内容提供了一种制备上文所述的组合物的方法。所述方法包括:(a)提供多个淋巴细胞;(b)将编码第一多肽链和第二多肽链的核酸分子引入多个淋巴细胞,以获得多个基因工程化的淋巴细胞;和(c)在细胞培养基中扩增多个基因工程化的淋巴细胞。
在一些实施方式中,所述方法可以包括:(a)提供多个淋巴细胞;(b)分别将编码第一多肽链和第二多肽链的第一核酸和第二核酸引入多个淋巴细胞,从而获得多个基因工程化的淋巴细胞;和(c)在细胞培养基中扩增多个基因工程化的淋巴细胞。在一些实施方式中,所述方法可以另外包括在引入第一核酸的步骤之后在细胞培养基中扩增多个第一淋巴细胞,或者在引入第二核酸的步骤之后在细胞培养基中扩增多个第二淋巴细胞。
可以在体外或离体进行获得上述淋巴细胞的肿瘤特异性基因修饰子集的组合物的方法。更特定形式的方法可以如PCT/EP2018/080343中所公开的,其内容通过引用整体并入本文。
术语“培养”或“扩增”是指在细胞可以增殖并避免衰老的条件下维持或培养细胞。例如,可以在任选地包含一种或多种生长因子(即,生长因子混合物)的培养基中培养细胞。在一些实施方式中,细胞培养基是限定的细胞培养基。细胞培养基可以包含新抗原肽。可以建立稳定的细胞系以允许细胞持续繁殖。
淋巴细胞
在淋巴细胞的扩增和基因修饰之前,获得来自受试者的淋巴细胞来源。淋巴细胞可以来自几个来源,包括外周血单核细胞、骨髓、淋巴结组织、脐带血、胸腺组织、来自感染部位组织、腹水、胸腔积液、脾组织和肿瘤。如本文所述,可以使用本领域可用的任何数量的淋巴细胞系。可以使用本领域技术人员已知的多种技术(如FicollTM分离),从在受试者中收集的单位血液中获得淋巴细胞。个体的循环血细胞可以通过单采血液分离术(apheresis)获得。血液分离术产品通常包含淋巴细胞,包括T淋巴细胞、单核细胞、粒细胞、B淋巴细胞,其他有核白细胞,红细胞和血小板。可以洗涤通过单采血液分离术收集的细胞以除去血浆级分,并将细胞放置在适宜的缓冲液或培养基中以用于后续处理步骤。可以用PBS洗涤细胞。或者,洗涤溶液可能不含钙和不含镁,或者可能不含很多(如果不是全部的话)二价阳离子。本领域普通技术人员将意识到的是,可以通过本领域技术人员公知的方法来完成洗涤步骤,如根据生产厂商的说明书使用半自动连续流离心机(例如,Cobe 2991细胞处理器,the Baxter CytoMate或e1Haemonetics Cell Saver 5)。洗涤后,可以将细胞重悬在各种生物相容性缓冲液中,比如例如,不含Ca2+、不含Mg2+PBS、PlasmaLyte A或含有或不含缓冲剂的其他盐溶液。或者,可以除去单采血液分离术样品中不需要的成分,并将细胞直接重悬在培养基中。
如本文所述,例如,通过PERCOLL梯度离心或逆流离心淘析,通过裂解红细胞和耗竭单核细胞从外周血中分离淋巴细胞。如有需要,可以通过阳性或阴性选择技术进一步分离特定亚群的淋巴细胞,如T淋巴细胞(即,Cd3+、CD28+、CD4+、CD8+、CD45RA+或CD45RO+T淋巴细胞)。例如,可以通过使用缀合的抗CD3/抗CD28小珠(即,3x28),如DYNABEADS M-450CD3/CD28 T,孵育足够长的时间(即,30分钟至24小时)分离T淋巴细胞,以阳性选择所需的T淋巴细胞。对于从白血病患者中分离T淋巴细胞,使用更长的孵育时间(如24小时)能够提高细胞性能。在与其他细胞类型相比T淋巴细胞很少的任何情况下,如从肿瘤组织或免疫受损的个体中分离TIL的任何情况下,都可以使用更长的孵育时间来分离T淋巴细胞。本领域技术人员将意识到,也可以使用多轮选择。进行选择程序并在活化和扩增过程中使用“未选择的”细胞可能是理想的。也可以对“未选择的”细胞进行新一轮选择。
可以通过针对阴性选择细胞的独特表面标记物的抗体组合来进行阴性选择富集淋巴细胞(例如,T淋巴细胞)群。一种方法是通过使用针对存在于阴性选择细胞中的细胞表面标记物的单克隆抗体混合物,通过阴性磁免疫粘附或流式细胞术对细胞进行分选和/或选择。例如,为了通过阴性选择富集CD4+细胞,单克隆抗体通常包括针对CD14、CD20、CD11b、CD16、HLA-DR和CD8的抗体。或者,通过抗C25缀合物小珠或其他相似的选择方法耗竭调节性T淋巴细胞。
还可以在洗涤步骤之后冷冻用于刺激的淋巴细胞。不希望受到理论的束缚,冷冻和接下来的解冻步骤通过消除细胞群中的粒细胞和在某种程度上单核细胞提供了更加均匀的产物。在除去血浆和血小板的洗涤步骤之后,可以将细胞悬浮在冷冻溶液中。尽管很多溶液和冷冻参数是本领域公知的并且在该背景下将是有用的,但是一种方法涉及使用含有20%DMSO和8%人血清白蛋白的PBS,或者含有10%葡聚糖40和5%右旋糖人白蛋白和和7.5%DMSO或31.25%血浆A、31.25%右旋糖5%、0.45%NaCl、10%葡聚糖40和5%右旋糖、20%人白蛋白血清和7.5%DMSO的培养基或其他适宜的细胞冷冻培养基,含有例如Hespan和PlasmaLyte A。然后,可以将细胞以每分钟1℃的速率在-80℃下冷冻,并储存在液氮储存罐的气相中。可以使用其他控制冷冻的方法,以及立即在-20℃或在液氮中进行不受控制的冷冻。
可以如本文所述将冷冻细胞解冻和洗涤,并在室温下静置一小时,然后使用本发明的方法进行活化。如本文所述,淋巴细胞可以扩增、冷冻并在以后使用。如本文所述,可以在如本文所述的特定疾病诊断之后不久,但在进行任何治疗之前,从患者中收集样品。可以在任何数量的相关治疗方式之前,从受试者的血液样品或单采血液分离术中分离细胞,包括但不限于使用药剂(如那他珠单抗、依法珠单抗)、抗病毒剂、化学疗法、射线、免疫抑制剂(如环孢菌素、硫唑嘌呤、甲氨蝶呤、霉酚酸酯和FK506)、抗体或其他免疫消融剂(如CAMPATH、抗CD3抗体、cytoxane、氟达拉滨、环孢菌素、FK506、雷帕霉素、霉酚酸、类固醇、FR901228和辐射)。这些药物抑制钙依赖性钙调神经磷酸酶(例如,环孢菌素和FK506)或抑制p70S6激酶,该酶对由生长因子(雷帕霉素)诱导的信号传导是重要的(Liu等,Cell 66:807-815,1991;Henderson等,Immun 73:316-321,199,Bierer等,Curr.Opin.Immun.,5:763-773,1993)。可以从患者中分离细胞并冷冻,以供随后与骨髓或干细胞移植、使用化学治疗剂(如氟达拉滨)、放疗外照射(XRT)、环磷酰胺或抗体(如OKT3或CAMPATH)的T淋巴细胞消融疗法一起(例如,在之前、同时或之后)使用。如本文所述,可以在之前分离细胞,并且可以冷冻以用于在B淋巴细胞消融疗法(如与CD20反应的药剂,例如,美罗华)后的治疗。
在对淋巴细胞进行基因修饰以表达所需的转基因之前或之后,通常可以使用诸如例如在美国专利6,352,694;6,534,055;6,905,680;6,692,964;5,858,358;6,887,466;6,905,681;7,144,575;7,067,318;7,172,869;7,232,566;7,175,843;5,883,223;6,905,874;6,797,514;6,867,041;和美国专利申请20060121005中描述的那些方法活化和扩增淋巴细胞。
载体
可以使用各种方法将转基因引入淋巴样细胞。这些方法包括但不限于使用具有整合能力的γ逆转录病毒或慢病毒和DNA转座对细胞进行转导。
可以将多种载体用于转基因的表达。某些病毒通过受体介导的内吞作用感染细胞或进入细胞,并整合入宿主细胞基因组并稳定有效地表达病毒基因的能力,使其成为将外来核酸转移到细胞中的引人注目的候选物。因此,在某些实施方式中,病毒载体用于将编码一个或多个转基因或其片段的核苷酸序列引入宿主细胞中进行表达。病毒载体可以包含编码与一个或多个控制序列(例如,启动子)可操作地连接的一个或多个转基因或其片段的核苷酸序列。或者,病毒载体可以不包含控制序列,而是依赖宿主细胞内的控制序列来驱动转基因或其片段的表达。可以用于递送核酸的病毒载体的非限制性实例包括腺病毒载体、AAV载体和逆转录病毒载体。
例如,腺相关病毒(AAV)可用于将编码一个或多个转基因或其片段的核苷酸序列引入宿主细胞中进行表达。此前已描述了AAV系统,并且其在本领域中通常是众所周知的(Kelleher和Vos,Biotechniques,17(6):1110-7,1994;Cotton等,Proc Natl Acad SciUSA,89(13):6094-6098,1992;Curiel,Nat Immun,13(2-3):141-64,1994;Muzyczka,CurrTop Microbiol Immunol,158:97-129,1992)。rAAV载体的生产和使用的细节在例如美国专利号5,139,941和4,797,368中进行了描述,其均出于所有目的通过引用整体并入本文。
在一些实施方式中,逆转录病毒表达载体可用于将编码一个或多个转基因或其片段的核苷酸序列引入宿主细胞中进行表达。此前已描述了这些系统,并且其在本领域中通常是众所周知的(Nicolas和Rubinstein,In,Rodriguez和Denhardt编著,Stoneham:Butterworth,pp.494-513,1988;Temin,In:Gene Transfer,Kucherlapati(ed.),NewYork:Plenum Press,pp.149-188,1986)。用于在哺乳动物细胞中进行真核表达的载体的实例包括AD5、pSVL、pCMV、pRc/RSV、pcDNA3、pBPV等,以及衍生自病毒系统(如牛痘病毒、腺相关病毒、疱疹病毒、逆转录病毒等)使用启动子(如CMV、SV40、EF-1、UbC、RSV、ADV、BPV和β-肌动蛋白)的载体。
逆转录病毒和适合包装细胞系的组合也可以找到用途,其中衣壳蛋白将具有感染靶细胞的功能。通常,将细胞和病毒在培养基中孵育至少约24小时。然后,在分析之前,在一些应用中,允许细胞在培养基中生长短时间(例如,24-73小时)或至少两周,以及可以允许细胞生长五周或更长时间。常用的逆转录病毒载体是“有缺陷的”,即不能产生生产性感染所需的病毒蛋白。载体的复制需要在包装细胞系中生长。逆转录病毒的宿主细胞特异性由包膜蛋白env(pl20)确定。包膜蛋白是由包装细胞系提供的。包膜蛋白至少有三种类型,亲嗜性、两性和异嗜性。与亲嗜性包膜蛋白一起包装的逆转录病毒(例如,MMLV)能够感染大多数小鼠和大鼠细胞类型。异嗜性包装细胞系包括BOSC23。携带两性包膜蛋白的逆转录病毒(例如,4070A)能够感染大多数哺乳动物细胞类型,包括人、犬和小鼠。两性包装细胞系包括PA12和PA317。与异嗜性包膜蛋白一起包装的逆转录病毒(例如,AKR env)能够感染大多数哺乳动物细胞类型,但小鼠细胞除外。载体可以包含必须随后例如使用重组酶系统(如Cre/Lox)除去的基因,或者表达其的细胞例如通过包含允许选择性毒性的基因(如疱疹病毒TK、BCL-xs等)而被破坏。适宜的诱导型启动子在所需靶细胞类型(即转染的细胞或其子代)中被激活。
有用的载体的非限制性实例包括逆转录病毒载体SFG.MCS和辅助质粒RD114,Peg-Pam3(Arber等,J Clin Invest 2015Jan 2;125(1):157-168)、慢病毒载体pRRL和辅助质粒R8.74和pMD2G(例如,Addgene质粒#12259)。在一些实施方式中,可以使用睡美人转座系统(Deniger等,2016Mol Ther.Jun;24(6):1078-1089)。在一些实施方式中,通过使其穿过一个小开口,使细胞变形,破坏细胞膜并使物质插入细胞中而将转基因引入细胞,例如,电穿孔(Xiaojun等,2017Protein Cell,8(7):514-526)或细胞方法。编码转基因的RNA的此类电穿孔方法可在细胞中瞬时表达此类转基因,从而限制工程化细胞的毒性和其他不良影响(Barrett等,2011Hum Gene Ther.Dec;22(12):1575-1586)。
用于将多核苷酸引入宿主细胞的物理方法包括磷酸钙沉淀、脂质转染、粒子轰击、显微注射、电穿孔等。用于产生包含外源载体和/或核酸的细胞的方法是本领域众所周知的。参见,例如,Sambrook等(2001,Molecular Cloning:A Laboratory Manual,ColdSpring Harbor Laboratory,New York)。
用于将多核苷酸引入宿主细胞的化学方法包括胶体分散系统,如大分子复合物、纳米胶囊、微球、小珠和基于脂质的系统,包括水包油乳剂、胶束、混合胶束和脂质体。用作体外和体内释放载剂的示例性胶体系统是脂质体(例如,人工膜囊泡)。
在其中使用非病毒递送系统的情况下,示例性递送载剂是脂质体。涉及将脂质制剂用于将核酸引入宿主细胞(体外、离体或体内)。在另一个方面中,核酸可以与脂质结合。可以将与脂质结合的核酸封装在脂质体的水性内部中,散布在脂质体的脂质双层内,通过与脂质体和寡核苷酸都结合的结合分子与脂质体结合,包裹在脂质体中,与脂质体形成复合物,分散在含有脂质的溶液中,与脂质混合,与脂质组合,作为混悬液包含在脂质、内容物中或与胶束的复合物中,或以其他方式与脂质结合。与脂质、脂质/DNA或脂质/表达载体结合的组合物不限于在溶液中的任何特定结构。例如,其可以胶束形式存在于双层结构中,也可以呈“坍塌”结构。其也可以简单地散布在溶液中,可能形成尺寸或形状不均匀的聚集体。脂质是脂肪物质,其可以是天然的或合成的脂质。例如,脂质包括天然存在于细胞质中的脂肪滴,以及一类含有长链脂肪族烃及其衍生物的化合物,如脂肪酸、醇、胺、氨基醇和醛。
可以从商业来源获得适于使用的脂质。例如,二肉豆蔻基磷脂酰胆碱(“DMPC”)可以从Sigma,St.Louis,MO获得;磷酸二十六烷基酯(“DCP”)可以从K&K Laboratories(Plainview,NY)获得;胆固醇(“Choi”)可以从Calbiochem-Behring获得;二肉豆蔻基磷脂酰甘油(“DMPG”)和其他脂质可以从Avanti Polar Lipids,Inc.(Birmingham,AL)获得。在氯仿或氯仿/甲醇中的脂质储备液可以储存在约-20℃下。将氯仿作为唯一溶剂,因为其比甲醇更容易蒸发。“脂质体”是一个通用术语,其包含通过产生双层或封闭的脂质聚集体而形成的各种独特的和多层的脂质载剂。脂质体的特征在于具有囊泡结构,该囊泡结构具有磷脂双层膜和内部水性介质。多层脂质体具有由水性介质所分隔的多个脂质层。当磷脂混悬在过量的水溶液中时,其会自发形成。脂质组分在形成封闭结构之前经历了自我重排,并在脂质双层之间捕获了溶解的水和溶质(Ghosh等,1991Glycobiology 5:505-10)。然而,还包括在溶液中具有与正常囊泡结构不同结构的组合物。例如,脂质可以具有胶束结构,也可以简单地以脂质分子的不均匀聚集体形式存在。还涉及脂质体(Lipofectamine)-核酸复合物。
无论用于将外源性核酸引入宿主细胞的方法如何,都可以通过一系列检测来确证在宿主细胞中存在重组DNA序列。此类测定包括,例如,本领域技术人员熟知的“分子生物学”测定,如Southern和Northern印迹、RT-PCR和PCR;生物化学测定,如检测是否存在特定肽,例如,通过免疫学方法(ELISA和Western印迹)或通过本文所述的测定以鉴定本发明范围内的药剂。
治疗方法
上文所述的药剂(例如,载体和细胞)可以用于治疗各种疾病的免疫疗法。因此,本公开内容还提供了一种治疗病症(如感染、癌症或肿瘤)的方法。所述方法包括向有需要的受试者施用治疗有效量的上文所述的组合物或药物组合物。
如本文所用,术语“受试者”和“患者”可以互换使用,而与受试者是否接受或当前正在接受任何形式的治疗无关。如本文所用,术语“受试者”和“受试者们”可以指任何脊椎动物,包括但不限于哺乳动物(例如,奶牛、猪、骆驼、美洲驼、马、山羊、家兔、绵羊、仓鼠、豚鼠、猫、犬、大鼠和小鼠、非人灵长类动物(例如,猴,如食蟹猴,黑猩猩等)和人)。受试者可以是人或非人。在更示例性的方面中,哺乳动物是人。在一些实施方式中,受试者是人。在一些实施方式中,受试者患有癌症。在一些实施方式中,受试者是免疫功能低下的。
癌症
在一些实施方式中,上文所述的药剂(例如,载体和细胞)可以用于治疗癌症或肿瘤的免疫疗法。如本文所用,“癌症”、“肿瘤”和“恶性肿瘤”均等同地涉及组织或器官的增生。如果组织是淋巴或免疫系统的一部分,则恶性细胞可以包括循环细胞的非实体瘤。其他组织或器官的恶性肿瘤可能会产生实体瘤。本发明的方法可以用于治疗淋巴细胞、循环免疫细胞和实体瘤。
可以治疗的癌症包括未血管化或基本上未血管化的肿瘤以及血管化的肿瘤。癌症可以包括非实体瘤(如血液系统肿瘤,例如,白血病和淋巴瘤)或者可以包括实体瘤。可以使用本发明的组合物治疗的癌症类型包括但不限于癌瘤、母细胞瘤和肉瘤,以及某些白血病或恶性淋巴样肿瘤、良性和恶性肿瘤和恶性肿瘤,例如,肉瘤、癌瘤和黑色素瘤。还包括成人肿瘤/癌症和小儿肿瘤/癌症。
血液系统癌症是血液或骨髓癌症。血液系统(或血源性)癌症的实例包括白血病,包括急性白血病(如急性淋巴细胞白血病、急性髓细胞白血病、急性髓性白血病、早幼粒细胞性、髓性单核细胞性、单核细胞性和红白血病)、慢性白血病(如慢性髓细胞性(粒细胞性)白血病、慢性髓性白血病和慢性淋巴细胞性白血病)、真性红细胞增多症、淋巴瘤、霍奇金氏病、非霍奇金氏淋巴瘤(惰性和高分级形式)、多发性骨髓瘤、华氏巨球蛋白血症、重链病、骨髓增生异常综合征、毛细胞白血病和骨髓增生异常。
实体瘤是通常不包含囊肿或液体区域的异常组织肿块。实体瘤可以是良性的或恶性的。不同类型的实体瘤以形成实体瘤的细胞类型命名(如肉瘤、癌瘤和淋巴瘤)。实体瘤(如肉瘤和癌瘤)的实例包括纤维肉瘤、粘液肉瘤、脂肪肉瘤、软骨肉瘤、骨肉瘤和其他肉瘤,滑膜、间皮瘤、尤因瘤、平滑肌肉瘤、横纹肌肉瘤、结肠癌、淋巴样恶性肿瘤、胰腺癌、乳腺癌、肺癌、卵巢癌、前列腺癌、肝细胞癌、鳞状细胞癌、基底细胞癌、腺癌、汗腺癌、甲状腺髓样癌、甲状腺乳头状癌、嗜铬细胞瘤的皮脂腺癌、乳头状癌、乳头状腺癌、髓样癌、支气管癌、肾细胞癌、肝癌、胆管癌、绒毛膜癌、威尔姆斯瘤、子宫颈癌、睾丸肿瘤、精原细胞瘤、膀胱癌、黑色素瘤和CNS肿瘤(如胶质瘤)(如脑干胶质瘤和混合性胶质瘤)、胶质母细胞瘤(也称为星形细胞瘤)、CNS淋巴瘤、生殖细胞瘤、髓母细胞瘤、神经鞘瘤、颅咽管瘤、室管膜瘤、松果体瘤、血管母细胞瘤、听神经瘤、少突胶质细胞瘤、脑膜瘤、神经母细胞瘤,视网膜母细胞瘤和脑转移。
还可以将上述药剂用于治疗感染性疾病的免疫治疗剂;例如,在使用识别感染性疾病抗原的CAR的程序中。因此,可以制备与多种形式的感染性疾病抗原结合的本文所述的多肽,从而在结合后诱导对感染性疾病抗原的免疫应答。本文所述的CAR可以设计为与之结合的感染性疾病抗原包括但不限于细菌抗原、病毒抗原、真菌抗原、寄生虫抗原和微生物毒素。在下文中更详细地考虑了每一类抗原的示例性形式。
细菌
本发明的多肽或CAR可以结合的细菌(特别是,其表位)的实例包括但不限于:铜绿假单胞菌(Pseudomonas aeruginosa)、荧光假单胞菌(Pseudomonas fluorescens)、嗜酸假单胞菌(Pseudomonas acidovorans)、产酸假单胞菌(Pseudomonas alcaligenes)、恶臭假单胞菌(Pseudomonas putida)、嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)、洋葱伯克霍尔德氏菌(Burkholderia cepacia)、嗜水气单胞菌(Aeromonas hydrophilia)、大肠杆菌(Escherichia coli)、氟氏柠檬酸杆菌(Citrobacter freundii)、鼠伤寒沙门氏菌(Salmonella enterica Typhimurium)、伤寒沙门氏菌(Salmonella enterica Typhi)、副伤寒沙门氏菌(Salmonella enterica Paratyphi)、肠炎沙门氏菌(Salmonella entericaEnteridtidis)、痢疾志贺氏菌(Shigella dysenteriae)、弗氏志贺氏菌(Shigellaflexneri)、宋内氏志贺氏菌(Shigella sonnei)、阴沟肠杆菌(Enterobacter cloacae)、产气肠杆菌(Enterobacter aerogenes)、肺炎克雷伯菌(Klebsiella pneumoniae)、产氧克雷伯菌(Klebsiella oxytoca)、粘质沙雷氏菌(Serratia marcescens)、土拉弗朗西斯菌(Francisella tularensis)、摩氏摩根菌(Morganella morganii)、变形杆菌(Proteusmirabilis)、寻常变形杆菌(Proteus vulgaris)、产碱普罗威登斯菌(Providenciaalcalifaciens)、雷氏普罗威登斯菌(Providencia rettgeri)、斯氏普罗威登斯菌(Providencia stuartii)、醋酸钙不动杆菌(Acinetobacter calcoaceticus)、溶血不动杆菌(Acinetobacter haemolyticus)、小肠结肠炎耶尔森氏菌(Yersinia enterocolitica)、鼠疫耶尔森氏菌(Yersinia pestis)、假结核耶尔森氏菌(Yersiniapseudotuberculosis)、中间耶尔森氏菌(Yersinia intermedia)、百日咳博德特氏菌(Bordetella pertussis)、副百日咳博德特氏菌(Bordetella parapertussis)、支气管败血博德特氏菌(Bordetella bronchiseptica)、流感嗜血杆菌(Haemophilus influenzae)、副流感嗜血杆菌(Haemophilus parainfluenzae)、溶血性嗜血杆菌(Haemophilushaemolyticus)、副溶血性嗜血杆菌(Haemophilus parahaemolyticus)、杜克雷嗜血杆菌(Haemophilus ducreyi)、多杀巴斯德氏菌(Pasteurella multocida)、溶血巴斯德氏菌(Pasteurella haemolytica)、卡他氏杆菌(Branhamella catarrhalis)、幽门螺杆菌(Helicobacter pylori)、弯曲杆菌(Campylobacter fetus)、空肠弯曲菌(Campylobacterjejuni)、大肠杆菌弯曲杆菌(Campylobacter coli)、伯氏疏螺旋体(Borreliaburgdorferi)、霍乱弧菌(Vibrio cholerae)、副溶血弧菌(Vibrio parahaemolyticus)、肺炎军团菌(Legionella pneumophila)、单核细胞增生李斯特氏菌(Listeriamonocytogenes)、淋病奈瑟氏球菌(Neisseria gonorrhoeae)、脑膜炎奈瑟氏球菌(Neisseria meningitidis)、阴道加德纳氏菌(Gardnerella vaginalis)、脆弱拟杆菌(Bacteroides fragilis)、狄氏拟杆菌(Bacteroides distasonis)、拟杆菌(Bacteroides)3452A同源群、普通拟杆菌(Bacteroides vulgatus)、卵形拟杆菌(Bacteroides ovalus)、多形拟杆菌(Bacteroides thetaiotaomicron)、单形拟杆菌(Bacteroides uniformis)、埃氏拟杆菌(Bacteroides eggerthii)、内生拟杆菌(Bacteroides splanchnicus)、艰难梭状芽孢杆菌(Clostridium difficile)、结核分枝杆菌(Mycobacterium tuberculosis)、鸟分枝杆菌(Mycobacterium avium)、细胞内分枝杆菌(Mycobacterium intracellulare)、麻风分枝杆菌(Mycobacterium leprae)、白喉棒状杆菌(Corynebacterium diphtheriae)、溃疡杆菌(Corynebacterium ulcerans)、肺炎链球菌(Streptococcus pneumoniae)、无乳链球菌(Streptococcus agalactiae)、化脓性链球菌(Streptococcus pyogenes)、粪肠球菌(Enterococcus faecalis)、屎肠球菌(Enterococcus faecium)、金黄色葡萄球菌(Staphylococcus aureus)、表皮葡萄球菌(Staphylococcus epidermidis)、腐生葡萄球菌(Staphylococcus saprophyticus)、中间葡萄球菌(Staphylococcus intermedius)、金黄色葡萄球菌猪葡萄球菌亚种(Staphylococcus hyicus subsp.hyicus)、溶血性葡萄球菌(Staphylococcus haemolyticus)、人型葡萄球菌(Staphylococcus hominis)和解糖葡萄球菌(Staphylococcus saccharolyticus)。在一个特定实施方式中,本发明的构建体包含与葡萄球菌蛋白A结合的结合分子。
病毒
本发明的多肽或CAR可以结合的病毒(或其表位)的实例包括但不限于:多瘤病毒JC(JCV)、I型人免疫缺陷病毒(HIV I)、乙型肝炎病毒(HBV)、丙型肝炎病毒(HCV)、巨细胞病毒(CMV)、爱泼斯坦巴尔病毒(EBV)、流感病毒血凝素(Genbank登录号J02132;Air,1981,Proc.Natl.Acad.Sci.USA 78:7639-7643;Newton等,1983,Virology 128:495-501)、人呼吸道合胞病毒G糖蛋白(Genbank登录号Z33429;Garcia等,1994,J.Virol.;Collins等,1984,Proc.Natl.Acad.Sci.USA 81:7683)、麻疹病毒血凝素(Genbank登录号M81899;Rota等,1992,Virology 188:135-142)、2型单纯疱疹病毒糖蛋白gB(Genbank登录号M14923;Bzik等,1986,Virology 155:322-333)、脊髓灰质炎病毒I VP1(Emini等,1983,Nature304:699)、HIV I的包膜糖蛋白(Putney等,1986,Science 234:1392-1395)、乙型肝炎病毒表面抗原(Itoh等,1986,Nature 308:19;Neurath等,1986,Vaccine 4:34)、白喉毒素(Audibert等,1981,Nature 289:543)、链球菌24M表位(Beachey,1985,Adv.Exp.Med.Biol.185:193)、淋球菌菌毛蛋白(Rothbard和Schoolnik,1985,Adv.Exp.Med.Biol.185:247)、伪狂犬病病毒g50(gpD)、伪狂犬病病毒II(gpB)、伪狂犬病病毒gIII(gpC)、伪狂犬病病毒糖蛋白H、伪狂犬病病毒糖蛋白E、传染性胃肠炎糖蛋白195、传染性胃肠炎基质蛋白、猪轮状病毒糖蛋白38、猪细小病毒衣壳蛋白、猪痢疾螺旋体保护性抗原、牛病毒性腹泻糖蛋白55、新城疫病毒血凝素神经氨酸酶、猪流感血凝素、猪流感神经氨酸酶、口蹄疫病毒、猪结肠病毒、猪流感病毒、非洲猪瘟病毒、猪肺炎支原体、传染性牛鼻气管炎病毒(例如,传染性牛鼻气管炎病毒糖蛋白E或糖蛋白G)或传染性喉气管炎病毒(例如,传染性喉气管炎病毒糖蛋白G或糖蛋白I)、拉克罗斯病毒的糖蛋白(Gonzales Scarano等,1982,Virology 120:42)、新生牛犊腹泻病毒(Matsuno和Inouye,1983,Infection andImmunity 39:155)、委内瑞拉马脑炎病毒(Mathews和Roehrig,1982,J.Immunol.129:2763)、庞塔托鲁病毒(Dalrymple等,1981,Replication of Negative Strand Viruses,Bishop and Compans(eds.),Elsevier,N.Y.,p.167)、小鼠白血病病毒(Steeves等,1974,J.Virol.14:187)、小鼠乳房肿瘤病毒(Massey和Schochetman,1981,Virology 115:20)、乙型肝炎病毒核心蛋白和/或乙型肝炎病毒表面抗原或者其片段或衍生物(参见,例如,1980年06月04日公开的英国专利公开号GB 2034323A;Ganem和Varmus,1987,Ann.Rev.Biochem.56:651 693;Tiollais等,1985,Nature 317:489 495)、马流感病毒或马疱疹病毒的抗原(例如,甲型马流感病毒/阿拉斯加91神经氨酸酶、甲型马流感病毒/迈阿密63神经氨酸酶、甲型马流感病毒/肯塔基81神经氨酸酶、1型马疱疹病毒糖蛋白B和I型马疱疹病毒糖蛋白D)、牛呼吸道合胞病毒或牛副流感病毒的抗原(例如,牛呼吸道合胞病毒附着蛋白(BRSV G)、牛呼吸道合胞病毒融合蛋白(BRSV F)、牛呼吸道合胞病毒核衣壳蛋白(BRSVN)、3型牛副流感病毒融合蛋白、3型牛副流感病毒血凝素神经氨酸酶)、牛病毒性腹泻病毒糖蛋白48或糖蛋白53、甲型肝炎、流感、水痘、腺病毒、I型单纯疱疹病毒(HSV I)、II型单纯疱疹病毒(HSV II)、牛瘟、鼻病毒、回声病毒、轮状病毒、呼吸道合胞病毒、乳头瘤病毒、巴氏病毒、棘皮病毒、虫媒病毒、汉坦病毒、柯萨奇病毒、腮腺炎病毒、麻疹病毒、风疹病毒、脊髓灰质炎病毒、II型人免疫缺陷病毒(HIV II)、任何小核糖核酸病毒科、肠病毒、杯状病毒科、任何诺沃克病毒群、囊膜病毒,如α病毒、黄病毒、冠状病毒、狂犬病病毒、马尔堡病毒、埃博拉病毒、副流感病毒、正粘病毒、布尼亚病毒、沙粒病毒、呼肠孤病毒、轮状病毒、环状病毒、I型人T细胞白血病病毒、II型人T细胞白血病病毒、猿猴免疫缺陷病毒、慢病毒、多瘤病毒、细小病毒、人疱疹病毒6、宫颈上皮性疱疹病毒I(B病毒)和痘病毒。
在某些实施方式中,本发明的多肽或CAR与HIV结合,在向其施用了病毒载体的受试者中诱导对所述病毒的免疫应答。HIV的各种抗原域(例如,表位)是本领域公知的,并且此类域包括结构性域,如Gag、Gag-聚合酶、Gag-蛋白酶、逆转录酶(RT)、整合酶(IN)和Env。HIV的结构性域通常进一步细分为多肽,例如,p55、p24、p6(Gag);p160、p10、p15、p31、p65(pol、prot、RT和IN);gp160、gp120和gp41(Ems)或Ogp140,如Chiron Corporation所构建的。此类多肽的分子变体也可以被本发明的多肽或CAR靶向结合,例如,诸如在PCT/US99/31245、PCT/US99/31273和PCT/US99/31272中描述的那些变体。
真菌
本发明的多肽或CAR可以结合的真菌(或其表位)的实例包括但不限于来自毛霉属、念珠菌属和曲霉属的真菌,例如,总状毛霉(Mucor racmeosus)、白色念珠菌(Candidaalbicans)和黑曲霉菌(Aspergillus niger)。
寄生虫
本发明的多肽或CAR可以结合的寄生虫(或其表位)的实例包括但不限于:弓形虫(Toxoplasma gondii)、苍白螺旋体(Treponema pallidun)、疟疾(Malaria)和隐孢子虫(Cryptosporidium)。
微生物毒素
本发明的多肽或CAR可以结合的微生物毒素(或其表位)的实例包括但不限于:由炭疽芽孢杆菌(Bacillus anthracis)、蜡状芽孢杆菌(Bacillus cereus)、百日咳博德特氏菌(Bordatella pertussis)、肉毒梭菌(Clostridium botulinum)、产气荚膜梭菌(Clostridium perfringens)、破伤风梭菌(Clostridium tetani)、白喉杆菌(Croynebacterium diptheriae)、Salmonella sp.、Shigella sp.、Staphyloccus sp.和霍乱弧菌(Vibrio cholerae)产生的毒素。诸如来自刀豆的蓖麻毒素和其他天然存在(例如,由生物体产生)的毒素和人工制备的毒素或其部分也可以与本发明的多肽或CAR结合。
可以以适当方法向待治疗(或预防)的疾病施用药物组合物。尽管可以通过临床试验确定适当的剂量,但是施用的量和频率将由诸如患者的状况以及患者疾病的类型和严重程度等因素来确定。
当指示“免疫学有效量”、“有效抗肿瘤的量”、“有效肿瘤抑制量”或“治疗量”时,待施用的本发明化合物的精确量可以由医师根据年龄、体重、肿瘤尺寸、感染或转移的程度和患者状况(受试者)的个体差异确定。通常可以指出的是,可以以104至109个细胞/kg体重(例如,105至106个细胞/kg体重)的剂量施用包含本文所述的淋巴细胞的药物组合物,包括在这些间隔内的所有整数值。淋巴细胞组合物也可以以这些剂量施用若干次。可以使用输注技术施用细胞是免疫疗法的普遍认知(参见,例如,Rosenberg等,New Eng.J.of Med.319:1676,1988)。通过监测患者的疾病体征并相应地调整治疗,医学领域的技术人员可以容易地确定针对特定患者的最佳剂量和治疗方案。
本发明组合物的施用可以以任何方便的方式进行,包括输注或注射(即,静脉内、鞘内、肌内、腔内、气管内、腹膜内或皮下)、透皮施用或本领域公知的其他方法。可以每两周一次、每周一次或更频繁地施用,但是在疾病或病症维持期可以降低频率。在一些实施方式中,通过静脉内输注施用组合物。
在某些情况下,将使用本文所述的方法或本领域公知的其他方法活化和扩增细胞(其中将淋巴细胞扩增至治疗水平)与任何数量的相关治疗方式一起向患者施用(例如,之前、同时或之后)。本文还描述了可以将淋巴细胞与化学疗法、射线、免疫抑制剂,如环孢菌素、硫唑嘌呤、甲氨蝶呤和FK506抗体、或其他免疫消融剂,如CAMPATH、抗癌抗体联用。CD3或其他抗体疗法、cytoxine、氟达拉滨、环孢菌素、FK506、雷帕霉素、霉酚酸、类固醇、FR901228、细胞因子和辐射。
本发明的组合物还可以与骨髓移植(例如,之前、同时或之后),使用化疗剂(如氟达拉滨)、放疗外照射(XRT)、环磷酰胺或抗体(如OKT3或CAMPATH)的T淋巴细胞消融疗法一起向患者施用。本文还描述了所述组合物可以在B淋巴细胞的消融疗法(如与CD20反应的药剂,例如,美罗华)之后施用。例如,受试者可以接受高剂量化学疗法的标准治疗,然后进行外周血干细胞移植。在某些情况下,在移植后,受试者接受扩增的淋巴细胞输注,或者在手术之前或之后施用扩增的淋巴细胞。
在一些实施方式中,所述方法还可以包括向受试者施用第二治疗剂。所述第二治疗剂是抗癌或抗肿瘤剂。在一些实施方式中,组合物在所述第二治疗剂(包括化学治疗剂和免疫治疗剂)之前、之后或同时向受试者施用。
在一些实施方式中,所述方法还包括施用治疗有效量的免疫检查点调节剂。免疫检查点调节剂的实例可以包括PD1、PDL1、CTLA4、TIM3、LAG3和TRAIL。所述检查点调节剂可以与本发明的组合物同时(simultaneously)、分别或并行(concurrently)施用。
“化学治疗剂”是用于治疗癌症的化学化合物。化学治疗剂的实例包括烷化剂,如塞替派和环磷酰胺(CYTOXANTM);烷基磺酸酯,如白消安、英丙舒凡和哌泊舒凡;氮丙啶,如苯并多巴、碳醌、甲基多巴和乌拉多巴(uredopa);乙亚胺和甲基蜜胺,包括六甲蜜胺、三亚乙基三聚氰胺、三亚乙基磷酰胺、三亚乙基硫代磷酰胺和三甲基三聚氰胺;产乙酸素(特别是布雷他辛和布雷他辛酮);喜树碱(包括合成类似物拓扑替康);苔藓抑素;愈伤他汀;CC-1065(包括其合成类似物阿多来新、卡折来新、比折来新);念珠藻素(特别是念珠藻素1和念珠藻素8);尾海兔素;杜卡霉素(包括合成类似物KW-2189和CBI-TMI);五加素;水鬼蕉碱;匍枝珊瑚醇;海绵抑素;氮芥,如苯丁酸氮芥、氯苯那嗪、氯磷酰胺、雌莫司汀、异环磷酰胺、甲氯乙胺、盐酸甲氧乙胺、美法仑、新霉素、酚非那西汀、泼尼松汀、曲磷酰胺、乌拉莫司汀;亚硝基脲,如卡莫司汀、氯唑霉素、铁莫司汀、洛莫斯汀、尼莫斯汀、拉尼莫司汀;抗生素,如烯二炔抗生素(例如,加利车霉素,参见,例如,Agnew Chem.Intl.Ed.Engl.33:183-186(1994);达尼米星,包括达尼米星A;埃司帕米星;以及新卡司他汀发色团和相关色蛋白烯二炔抗生素发色团)、阿克拉霉素、放线菌素、安曲霉素、重氮丝氨酸、博来霉素、卡奇霉素、卡柔比星、洋红霉素、嗜癌素、色霉素、更生霉素、柔红霉素、地托比星、6-重氮-5-氧代-L-正亮氨酸、阿霉素(包括吗啉代-阿霉素、氰基吗啉代-阿霉素、2-吡咯啉-阿霉素和脱氧阿霉素)、表柔比星、埃索比星、伊达比星、麻西罗霉素、丝裂霉素、霉酚酸、线霉素、寡霉素、培洛霉素、泛霉素、嘌呤霉素、奎霉素、罗多比星、链黑霉素、链脲菌素、杀结核菌素、乌苯美司、净司他丁、佐柔比星;抗代谢物,如甲氨蝶呤和5-氟尿嘧啶(5-FU);也算类似物,如二甲叶酸、甲氨蝶呤、蝶罗呤、三甲曲沙;嘌呤类似物如氟达拉滨、6-巯嘌呤、硫咪嘌呤、硫鸟嘌呤;嘧啶类似物如安西他滨、阿扎胞苷、6-阿扎尿苷、卡莫氟、阿糖胞苷、二脱氧尿苷、去氧氟尿苷、依诺他滨、氟尿苷、5-FU;雄激素类如卡普睾酮、屈他雄酮丙酸酯、环硫雄醇、美雄烷、睾内酯;抗肾上腺类如氨鲁米特、米托坦、曲洛司坦;叶酸补充剂如叶酸;醋葡醛内酯;醛磷酰胺糖苷;氨基乙酰乙酸;安丫啶;比曲比新;比生群;依达曲沙;地佛法明;脱羰秋水仙碱;地吖醌;依氟鸟氨酸;依利醋铵;埃博霉素;依托格鲁;硝酸镓;羟基脲;香菇多糖;氯尼达明;美登木素生物碱,如美登素和安托霉素;米托胍腙;米托蒽醌;莫哌达醇;硝氨丙吖啶;喷司他丁;苯来美特;吡柔比星;鬼臼酸;2-乙基酰肼;丙卡巴嗪;雷佐生;利索新;西左非兰;锗螺胺;替奴佐酸;三亚胺醌;2,2’,2”-三氯三乙胺;毛霉菌素(特别是T-2毒素、维拉库林A、罗丹蛋白A和胍基啶);乌拉坦;长春地辛;达卡巴嗪;甘露莫司汀;二溴甘露醇;二溴卫矛醇;哌泊溴烷;加西托新;阿糖胞苷(“Ara-C”);环磷酰胺;塞替派;紫杉烷类,例如,紫杉醇和多西他赛苯丁酸氮芥;吉西他滨;6-硫鸟嘌呤;巯基嘌呤;甲氨蝶呤;铂类似物,如顺铂和卡铂;长春碱;铂;依托泊苷(VP-16);异环磷酰胺;撕裂霉素C;米托蒽醌;长春新碱;长春瑞滨;纳维滨、诺万隆;替尼泊苷;道诺霉素;氨基蝶呤;希罗达;伊班膦酸;CPT-11;拓扑异构酶抑制剂RFS 2000;二氟甲基鸟氨酸(DMFO);视黄酸;卡培他滨;以及上述任何的药学上可接受的盐、酸或衍生物。在该定义中还包括用于调节或抑制激素对肿瘤的作用的抗激素药,如抗雌激素,包括例如他莫昔芬、雷洛昔芬,芳香化酶抑制剂4(5)-咪唑、4-羟基他莫昔芬、三噁昔芬、酮洛芬、LY117018、奥那司酮和托瑞米芬(法乐通);和抗雄激素,如氟他胺、尼鲁米特、比卡鲁胺、亮丙瑞林、希罗达、吉西他滨、KRAS突变共价抑制剂和戈舍瑞林;以及上述任何的药学上可接受的盐、酸或衍生物。其他实例包括伊立替康、奥沙利铂和其他标准结肠癌方案。
“免疫治疗剂”是用于治疗癌症的生物药剂。免疫治疗剂的实例包括阿特朱单抗、阿维单抗、博纳吐单抗、达雷木单抗、西米单抗、德瓦鲁单抗、埃罗妥珠单抗、laherparepvec、伊匹单抗、纳武单抗、阿托珠单抗、奥法木单抗、派姆单抗、西妥昔单抗和他莫基因。
在下述实例中,通过使用基于TCR的嵌合抗原受体工程化T细胞,所述T细胞在HLA-A0201的背景下对NY-ESO-1保持较高的亲和性和特异性,将上文所述的方法用于在HLA-A0201背景下增强对表达肿瘤相关抗原NY-ESO-1的癌症的过继免疫疗法的有效性。
NY-ESO-1是一种仅在胎儿和睾丸组织中正常表达的蛋白,但是在一些实体恶性肿瘤中异常表达。这将NY-ESO-1添加到了在种系中适当表达的分子列表中,并且被一些癌症异常表达。这些分子被称为“癌症/睾丸”抗原,可以将其用作抗原导向免疫疗法的靶点。癌-睾丸抗原NY-ESO-1在很多实体瘤中表达,在成熟的体细胞组织中表达受限,这使其成为肿瘤免疫疗法的非常有吸引力的靶点。使用工程化T细胞靶向NY-ESO-1在一些成人肿瘤的治疗中显示出临床有效性。
该方法涉及:(1)在CAR设计中整合了TCR共刺激信号传导元件;(2)通过使用简并密码子沉默突变外源性核酸序列之一来产生包含编码相同多肽序列的两个或更多个核酸序列的载体核酸序列,以减少两个核酸序列之间的同源性,同时保持编码的多肽序列;(3)整合人CD28、或人4-1BB、或者两者的TCR共刺激信号传导元件;和(4)在同一TCR-CAR构建体中包含相同或不同的跨膜域。还公开了此类CAR基因的特异性核酸序列。这种方法通过使用1G4 195LY TCR的α链和β链的EC域,并引入TCR信号传导元件CD3Z或CD3E以及共刺激元件CD28、4-1BB或CD28和4-1BB的组合制备得到的具有抗NY-ESO-1特异性的基于TCR的CAR,以通过TCR-CAR介导的刺激增强表达TCR-CAR的T细胞活性。这导致在患者中的抗肿瘤活性增加。
定义
T细胞受体或TCR是存在于T细胞或T淋巴细胞表面上的一种蛋白复合物,其负责将抗原片段识别为与MHC分子结合的肽。TCR和抗原肽之间的结合具有相对较低的亲和性并且是简并性的;即,很多TCR识别相同抗原肽和很多抗原肽被同一TCR识别。大多数T细胞都具有以多种蛋白复合物形式存在的TCR。TCR由两条不同的蛋白链组成。在人类中,95%的T细胞中的TCR由α(α)链和β(β)链组成(分别由TRA和TRB编码),而在5%的T细胞中TCR由γ和δ(γ/δ)链组成(分别由TRG和TRD编码)。该比例在发育过程中和患病状态(如白血病)下发生变化。在物种之间也有所不同。TCR的每条链均由两个胞外域组成:可变(V)区和恒定(C)区。恒定区靠近细胞膜,随后是跨膜区和短的胞质尾,而可变区与肽/MHC复合物结合。出于本发明的目的,术语“T细胞受体链的恒定区或其部分”还包括这样的实施方式,其中T细胞受体链的恒定区(从N末端到C末端)随后是跨膜区和胞质尾,这样跨膜区和胞质尾天然地与T细胞受体链的恒定区连接。
如本文所用,术语“抗原受体”或“抗原识别受体”指能够响应于抗原结合而激活免疫细胞(例如,T细胞)的受体。特别地,术语“抗原受体”包括工程化的受体,其赋予免疫效应细胞(如T细胞)任意特异性。根据本发明的抗原受体可以存在于T细胞上,例如,代替或补充T细胞自身的T细胞受体。此类T细胞不必为了识别靶细胞而需要处理和呈递抗原,而是可以优选地特异性识别存在于靶细胞上的任何抗原。优选地,所述抗原受体在细胞表面上表达。特别地,该术语包括人工或重组受体,其包含识别(即,结合至)靶细胞(例如,通过将抗原结合位点或抗原结合域与靶细胞表面表达的抗原结合)上的靶结构(例如,抗原)的并且可以赋予对在细胞表面上表达所述抗原受体的免疫效应细胞(如T细胞)的特异性的单一分子或分子的复合物。优选地,抗原受体对靶结构的识别导致表达所述抗原受体的免疫效应细胞的活化。抗原受体可以包含一个或多个蛋白单元,所述蛋白单元包含如本文所述的一个或多个域。术语“抗原受体”优选地不包括天然存在的T细胞受体。根据本发明,术语“抗原受体”优选与术语“嵌合抗原受体”、“嵌合T细胞受体”和“人工T细胞受体”同义。
示例性的抗原识别受体可以是天然的或基因工程化的TCR,或基因工程化的TCR样mAb(Hoydahl等,Antibodies 2019 8:32)或CAR,其中肿瘤抗原结合域与能够活化免疫细胞(例如,T细胞)的胞内信号传导域融合。此前已公开了表达针对特定癌症抗原的天然TCR的T细胞克隆(Traversari等,J Exp Med,1992 176:1453-7;Ottaviani等,Cancer ImmunolImmunother,2005 54:1214-20;Chaux等,J Immunol,1999 163:2928-36;Luiten和van derBruggen,Tissue Antigens,2000 55:149-52;van der Bruggen等,Eur J Immunol,199424:3038-43;Huang等,J Immunol,1999 162:6849-54;Ma等,Int J Cancer,2004 109:698-702;Ebert等,Cancer Res,2009 69:1046-54;Ayyoub等,J Immunol 2002 168:1717-22;Chaux等,European Journal of Immunology,2001 31:1910-16;Wang等,CancerImmunol Immunother,2007 56:807-18;Schultz等,Cancer Research,2000 60:6272-75;Cesson等,Cancer Immunol Immunother,2010 60:23-25;Zhang等,Journal ofImmunology,2003 171:219-25;Gnjatic等,PNAS,2003 100:8862-67;Chen等,PNAS,2004)。
术语“嵌合抗原受体”或“CAR”指一组多肽,在最简单的实施方式中通常为两个,其当在免疫效应细胞中时,提供了对靶细胞具有特异性和产生胞内信号的细胞。在一些实施方式中,CAR至少包含胞外抗原结合域、跨膜域和胞质信号传导域(在本文中也称为“胞内信号传导域”),其包含来源于如下文所定义的刺激分子和/或共刺激分子的功能性信号传导域。在一些实施方式中,该组多肽是在相同多肽链中,例如包含嵌合融合蛋白。在一些实施方式中,该组肽彼此之间是不连续的,例如,在不同多肽链中。在一些实施方式中,该多肽组包含二聚化开关,当存在二聚化分子时,所述二聚化开关能够使所述多肽彼此偶联,例如,能够将抗原结合域与胞内信号传导域偶联。在一个方面中,CAR的共刺激分子是与T细胞受体复合物(例如,CD3ζ)结合的ζ链。在一个方面中,胞质信号传导域包含主要信号传导域(例如,CD3-ζ的主要信号传导域)。在一个方面中,胞质信号传导域还包含一个或多个衍生自如下文所定义的至少一种共刺激分子的一个或多个功能性域。在一个方面中,所述共刺激分子是从本文所述的共刺激分子中选择的,例如,4-1BB(即,CD137)、CD27和/或CD28。
术语“基于TCR的CAR”或“TCR-CAR”指包含由TCRα、β、γ或δ链形成的抗原结合域或其抗原结合部分的CAR。
术语“刺激分子”指由免疫细胞(例如,T细胞、NK细胞或B细胞)表达的分子,其提供了针对免疫细胞信号传导途径的至少一些方面以刺激方式调控免疫细胞激活的一个或多个胞质信号传导序列。在一个方面中,所述信号是例如由TCR/CD3复合物与负载肽的MHC分子结合引发的主要信号,并且导致介导T细胞应答,包括但不限于,增殖、活化、分化等。以刺激方式起作用的主要胞质信号传导序列(也称为“主要信号传导域”)可以含有信号传导基序,将其称为基于免疫受体酪氨酸的激活基序或ITAM。在本发明中特别使用的包含ITAM的胞质信号传导序列的实例包括但不限于衍生自CD3ζ、常见FcRγ(FCER1G)、FcγRIIa、FcRβ(FcεR1b)、CD3γ、CD3Δ、CD3ε、CD79a、CD79b、DAP10和DAP12的那些。在本发明的特异性CAR中,本发明的任何一个或多个CAR中的胞内信号传导域包含胞内信号传导序列,例如,CD3-ζ的主要信号传导序列。在本发明的特异性CAR中,CD3-ζ的主要信号传导序列是本文提供的序列,或来自非人物种的等效残基,例如,小鼠、啮齿类动物、猴、类人猿等。在本发明的特异性CAR中,CD3-ζ的主要信号传导序列是如本文所提供的序列,或来自非人物种的等效残基,例如,小鼠、啮齿类动物、猴、类人猿等。
术语“共刺激分子”指在T细胞上与共刺激配体特异性结合的同源结合伴侣,从而介导T细胞的共刺激应答,比如但不限于增殖。共刺激分子是除抗原受体或其配体以外的细胞表面分子,其有助于有效的免疫应答。共刺激分子包括但不限于I类MHC分子、TNF受体蛋白、免疫球蛋白样蛋白、细胞因子受体、整联蛋白、信号传导淋巴细胞激活分子(SLAM蛋白)、活化NK细胞受体、BTLA、Toll配体受体、OX40、CD2、CD7、CD27、CD28、CD30、CD40、CDS、ICAM-1、LFA-1(CD11a/CD18)、4-1BB/CD137、B7-H3、CDS、ICAM-1、ICOS(CD278)、GITR、BAFFR、LIGHT、HVEM(LIGHTR)、KIRDS2、SLAMF7、NKp80(KLRF1)、NKp44、NKp30、NKp46、CD19、CD4、CD8α、CD8β、IL2Rβ、IL2Rγ、IL7Rα、ITGA4、VLA1、CD49a、ITGA4、IA4、CD49D、ITGA6、VLA-6、CD49f、ITGAD、CD11d、ITGAE、CD103、ITGAL、CD11a、LFA-1、ITGAM、CD11b、ITGAX、CD11c、ITGB1、CD29、ITGB2、CD18、LFA-1、ITGB7、NKG2D、NKG2C、TNFR2、TRANCE/RANKL、DNAM1(CD226)、SLAMF4(CD244、2B4)、CD84、CD96(Tactile)、CEACAM1、CRTAM、Ly9(CD229)、CD160(BY55)、PSGL1、CD100(SEMA4D)、CD69、SLAMF6(NTB-A、Ly108)、SLAM(SLAMF1、CD150、IPO-3)、BLAME(SLAMF8)、SELPLG(CD162)、LTBR、LAT、GADS、SLP-76、PAG/Cbp、CD19a和与CD83特异性结合的配体。
共刺激胞内信号传导域指共刺激分子的胞内部分。胞内信号传导域可以包含衍生其的分子的整个胞内部分或整个天然胞内信号传导域,或者其功能性片段或衍生物。
如本文所用,两个多肽(或核酸)序列是“基本上不同的”指两个序列彼此之间的同一性小于95%(例如,90%、85%、80%、75%、70%、65%、60%、55%或50%)。
如本文所用,术语“功能性变体”指与野生型具有基本上或显著序列同一性或相似性的修饰的多肽或蛋白或转基因,这种功能性变体保留了以其为变体的野生型多肽或蛋白或转基因的生物活性。在一些实施方式中,使用治疗性多肽或蛋白或转基因的功能性变体。
本发明公开的肽、多肽或蛋白的保守性修饰或功能性等同物指所述肽、多肽或蛋白的多肽衍生物,例如,具有一个或多个点突变、插入、缺失、截短、融合蛋白或其组合的蛋白。其基本上保留了母体肽、多肽或蛋白(如在发明中公开的那些)的活性。在通常情况下,保守性修饰或功能性等同物与母体(例如,本文公开的那些中的一个)至少60%(例如,60%和100%之间的任何数字,包括,例如,60%、70%、75%、80%、85%、90%、95%、96%、97%、98%和99%)相同。因此,在本发明的范围内,铰链区具有一个或多个点突变、插入、缺失、截短、融合蛋白或其组合。
在一些情况下,可以通过选择在维持(1)取代区域中肽骨架的结构,(b)在靶位点分子的电荷或疏水性;或(c)侧链体积方面其作用没有显著性差异的取代进行氨基酸取代。例如,基于侧链性质可以将天然存在的残基分为几类:(1)疏水性氨基酸(正亮氨酸、甲硫氨酸、丙氨酸、缬氨酸和异亮氨酸);(2)中性亲水性氨基酸(半胱氨酸、丝氨酸、苏氨酸、天冬酰胺和谷氨酰胺);(3)酸性氨基酸(天冬氨酸和谷氨酸);(4)碱性氨基酸(组氨酸、赖氨酸和精氨酸);(5)影响链取向的氨基酸(甘氨酸和脯氨酸);和(6)芳香族氨基酸(色氨酸、酪氨酸和苯丙氨酸)。在这些类别中进行的取代可以认为是保守性取代。取代的实例包括但不限于缬氨酸取代丙氨酸、赖氨酸取代精氨酸、谷氨酰胺取代天冬酰胺、谷氨酸取代天冬氨酸、丝氨酸取代半胱氨酸、天冬酰胺取代谷氨酰胺、天冬氨酸取代谷氨酸、脯氨酸取代甘氨酸、精氨酸取代组氨酸、亮氨酸取代异亮氨酸、异亮氨酸取代亮氨酸、精氨酸取代赖氨酸、亮氨酸取代甲硫氨酸、亮氨酸取代苯丙氨酸、甘氨酸取代脯氨酸、苏氨酸取代丝氨酸、丝氨酸取代苏氨酸、酪氨酸取代色氨酸、苯丙氨酸取代酪氨酸和/或亮氨酸取代缬氨酸。下表中显示了示例性取代。可以将氨基酸取代引入母体蛋白,并筛选保留母体蛋白生物学活性的产物。
原始残基
示例性取代
Ala(A)
Val;Leu;Ile
Arg(R)
Lys;Gln;Asn
Asn(N)
Gln;His;Asp,Lys;Arg
Asp(D)
Glu;Asn
Cys(C)
Ser;Ala
Gln(Q)
Asn;Glu
Glu(E)
Asp;Gln
Gly(G)
Ala
His(H)
Asn;Gln;Lys;Arg
Ile(I)
Leu;Val;Met;Ala;Phe;正亮氨酸
Leu(L)
正亮氨酸;Ile;Val;Met;Ala;Phe
Lys(K)
Arg;Gln;Asn
Met(M)
Leu;Phe;Ile
Phe(F)
Trp;Leu;Val;Ile;Ala;Tyr
Pro(P)
Ala
Ser(S)
Thr
Thr(T)
Val;Ser
Trp(W)
Tyr;Phe
Tyr(Y)
Trp;Phe;Thr;Ser
Val(V)
Ile;Leu;Met;Phe;Ala;正亮氨酸
如本文所用,两条氨基酸序列之间的同源性百分比等于两条序列之间的同一性百分比。两条序列之间的同一性百分比是所述序列共享的相同位置数的函数(即,%同源性=相同位置数/位置总数x 100),其中考虑了空位数,以及每个空位的长度,需要引入这些空位以实现两条序列的最佳比对。如以下非限制性实例中所述,可以使用数学算法来完成序列的比较和两条序列之间的同一性百分比的确定。
可以使用E.Meyers和W.Miller算法(Comput.Appl.Biosci.,4:11-17(1988))确定两条氨基酸序列之间的同一性百分比,已将所述算法整合至ALIGN程序(2.0版)中,使用PAM120残基权重表,空位长度罚分12和空位罚分4。此外,可以使用Needleman和Wunsch(J.Mol.Biol.48:444-453(1970))算法确定两条氨基酸序列之间的同一性百分比,已将所述算法整合至GCG软件包中的GAP程序(可以从www.gcg.com获得),使用Blossum 62矩阵或PAM250矩阵,空位权重16、14、12、10、8、6或4和长度权重1、2、3、4、5或6。
如本文所用,术语“抗体”不仅指完整抗体分子,而且指保留抗原结合能力的抗体分子的片段。此类片段也是本领域熟知的,并且在体外和体内均定期使用。因此,如本文所用,术语“抗体”不仅指完整免疫球蛋白分子,而且指众所周知的活性片段f(ab′)2和fab。F(ab′)2和缺少完整抗体Fe片段的fab片段,从循环中更迅速清除,并且与完整抗体的非特异性组织结合可能更少(Wahl等,J.Nucl.Med.24:316-325(1983))。本发明的抗体包括完整的天然抗体、双特异性抗体;嵌合抗体;fab、fab′、单链v区片段、融合多肽和非常规抗体。
如本文所用,术语“单链可变片段”或“scFv”是共价连接形成VH::VL异二聚体的免疫球蛋白的重链(VH)和轻链(VL)的可变区的融合蛋白。重链(VH)和轻链(VL)直接连接或通过肽编码接头(例如,10、15、20、25个氨基酸)连接,所述接头将VH的n末端与VL的C末端,或者将VH的C末端与VL的N末端连接。接头通常富含甘氨酸以提高柔性,以及富含丝氨酸或苏氨酸以提高溶解度。尽管去除恒定区并引入接头,scFv蛋白仍保留了原始免疫球蛋白的特异性。如Huston等(Proc.Nat.Acad.Sci.,85:5879-5883,1988)所描述的,单链Fv多肽抗体可以从包含VH和VL编码序列的核酸表达。亦参见,美国专利号5,091,513、5,132,405和4,956,778;和美国专利公开号20050196754和20050196754。已描述了具有抑制活性的拮抗性scFv(参见,例如,Zhao等,Hybridoma(Larchmont)2008 27(6):455-51;Peter等,Jcachexiasarcopenia muscle 2012Aug.12;Shieh等,J Immunol 2009183(4):2277-85;Giomarelli等,Thromb Haemost 2007 97(6):955-63;Fife等,J Clin Invst 2006 116(8):2252-61;Brocks等,Immunotechnology 19973(3):173-84;Moosmayer等,Ther Immunol 1995 2(10:31-40))。已描述了具有刺激活性的激动性scFv(参见,例如,Peter等,J Bioi Chern200325278(38):36740-7;Xie等,Nat Biotech 1997 15(8):768-71;Ledbetter等,CritRev Immunol 1997 17(5-6):427-55;Ho等,Biochim Biophys Acta 20031638(3):257-66)。
如本文所用,“治疗(treating)”或“治疗(treatment)”指向患有病症的受试者施用化合物或药剂,其目的是治愈、减轻、缓解、补救、延迟其发作、预防或改善所述病症、病症的症状、继发于所述病症的疾病状态或对所述病症的易感性。
“有效量”或“治疗有效量”指能够在治疗的受试者中产生医学上理想的结果的化合物或药剂的量。可以在体内或离体进行治疗方法,单用或与其他药物或疗法联用。治疗有效量可以以一次或多次施用、应用或剂量施用,并且并非旨在限于特定制剂或施用途径。
将术语“有效量”、“有效剂量(dose)”或“有效剂量(dosage)”定义为足以达到或至少部分达到所需效果的量。药物或治疗剂的“治疗有效量”或“治疗有效剂量”是指当单独使用或与另一种治疗剂组合使用时,可促进疾病消退的任何量的药物,所述疾病消退表现为疾病症状的严重程度降低,无疾病症状时期的频率和持续时间增加,或者预防由于疾病困扰而导致的功能障碍或残疾。药物的“预防有效量”或“预防有效剂量”是当单独施用或与另一种治疗剂组合施用于处于发生疾病或经历疾病复发风险的受试者以抑制所述疾病的发展或复发的药物的量。治疗或预防剂促进疾病消退或抑制所述疾病发展或复发的能力可以使用熟练技术人员公知的多种方法来评价,如在临床试验期间的人类受试者中,在预测在人体内有效性的动物模型系统中,或通过在体外测定中测定药剂的活性。
如本文所用,术语“药学上可接受的”指不消除组合物的生物学活性或性质且相对无毒的物质(如载体或稀释剂),即,可以将所述物质施用于个体而不会引起有害的生物学作用或以有害的方式与其中所含的所述组合物的任何组分相互作用。
术语“药学上可接受的载体”包括药学上可接受的盐、药学上可接受的物质、组合物或载体,如液体或固体填充剂、稀释剂、赋形剂、溶剂或封装物质,其参与在受试者内或向受试者携带或运输本发明的一种或多种化合物,以使得其可以执行其预期功能。通常,将此类化合物从一个器官或身体的一部分携带或运输到另一器官或身体的一部分。从与制剂与其他成分的相容性的意义上讲,每种盐或载体必须是“可接受的”,并且对受试者无害。可以作为药学上可接受的载体的物质的一些实例包括:糖,如乳糖、葡萄糖和蔗糖;淀粉,如玉米淀粉和马铃薯淀粉;纤维素及其衍生物,如羧甲基纤维素钠、乙基纤维素和乙酸纤维素;粉状西黄芪胶;麦芽;明胶;滑石粉;赋形剂,如可可脂和栓剂蜡;油,如花生油、棉籽油、红花油、芝麻油、橄榄油、玉米油和大豆油;二醇,如丙二醇;多元醇,如甘油、山梨糖醇、甘露醇和聚乙二醇;酯,如油酸乙酯和月桂酸乙酯;琼脂;缓冲剂,如氢氧化镁和氢氧化铝;海藻酸;无热原水;等渗盐水;林格氏溶液;乙醇;磷酸缓冲盐溶液;稀释剂;制粒剂;润滑剂;粘合剂;崩解剂;润湿剂;乳化剂;着色剂;脱模剂;包衣剂;甜味剂;调味剂;增香剂;防腐剂;抗氧化剂;增塑剂;稠化剂;增稠剂;硬化剂;固化剂;悬浮剂;表面活性剂;湿润剂;载体;稳定剂;和在药物制剂中使用的其他无毒相容性物质,或其任何组合。如本文所用,“药学上可接受的载体”还包括与化合物的活性相容且是受试者生理学上可接受的任何和所有包衣、抗菌和抗真菌剂以及吸收延迟剂等。也可以将补充活性化合物加入组合物中。
如本文所用,当应用于一个或多个值时,术语“约”或“大约”指类似于规定的参考值的值。在一些实施方式中,除非另有说明或从上下文可以明显看出(除非其中该数字将超出可能值的100%),术语“约”或“大约”指落入规定参考值的任一方向(大于或小于)内25%、20%、19%、18%、17%、16%、15%、14%、13%、12%、11%、10%、9%、8%、7%、6%、5%、4%、3%、2%、1%或以下之内的值的范围。除非本文另外指出,否则术语“约”旨在包括接近就单个成分、组合物或实施方式的功能而言是等效的所述范围的值(例如,重量百分比)。应当理解的是,无论在本文的何处提供数值和范围,这些数据和范围所涵盖的所有数值和范围都旨在涵盖在本发明的范围。此外,本申请还包括落入这些范围内的所有值以及值的范围的上限或下限。
实施例
实施例1:基于抗NY-ESO-1TCR-、抗NY-ESO-1TCR-CAR-、抗间皮素CAR-和GFP的MGF逆转录病毒载体的构建
逆转录病毒构建体的构建
为了在T细胞中组成性表达目标基因,最初使用Mulligan博士(图1C)开发的MFG逆转录病毒骨架和SFG逆转录病毒骨架(美国专利6,140,111)构建了编码目标基因的重组逆转录病毒载体,如特异性针对人NY-ESO-1衍生的肽/A2复合物的天然形式的TCR(NT1、NT1b)或TCR-CAR(NT2、NT3、NT4、NT5、NT6、NT21、NT22、NT23、NT24,NT25、NT26、NT27和T28)(图1A和图1B以及表1)。
表1:编码抗NY-ESO-1TCR、抗NY-ESO-1TCR-CAR、抗间皮素CAR和GFP的基因列表
将GFP作为标记物,以通过流式细胞术(FACS)和/或通过荧光显微镜追踪基于GFP的载体转导的T细胞。选择对人间皮素具有高亲和性的抗人间皮素sFv(GenBank ID:AF035617.1)(Chowdhury PS等,Proc Natl Acad Sci U S A.1998;95:669-74)用于构建基于抗间皮素的CAR。此前已在成功建立第二代和第三代功能性抗间皮素CAR中描述了抗间皮素sFv的这种用途(Carpenito C.等,Proc Natl Acad Sci U S A.2009;106:3360-5)。
核酸之间的重组是分子生物学中公认的现象。通常将需要参与的核酸序列之间具有较强序列同源性的遗传重组称为同源重组。尽管大多数基因敲除策略都采用同源重组来实现有针对性的敲除,但在某些系统中,基因重组的发生会不利地影响基因操作。特别地,同源重组事件可不利地影响载体,特别是病毒载体(例如,腺病毒、逆转录病毒、腺相关病毒、疱疹病毒等)的构建和产生,其中在例如病毒载体通过一个或多个宿主细胞和/生物体传代和/或增殖期间,在单个、稳定病毒载体内保持高度同源的序列(例如,相同多肽序列)而没有同源重组通常是理想的。
将此前描述的方法(美国专利号9,206,440)用于载体设计,以克服与核酸之间潜在的同源重组相关的此类问题,以使得能够在单个病毒载体上的单个蛋白分子中递送编码高度同源(例如,相同)多肽片段的两个或更多个核酸序列。在这种方法中,采用在核苷酸序列水平的沉默突变方法来产生病毒载体序列,所述病毒载体序列包含编码两个或更多个高度同源(例如,相同)多肽或其多肽域的核酸序列,但在此类核酸序列之间具有降低的同源重组风险,甚至在例如在宿主细胞中延长传代和/或多重感染、染色体整合和/或切除事件中也是如此。
此前已描述了(Robbins PF等,J Clin Oncol.2011;29:917-24;和Robbins PF等,J Immunol.2008;180:6116–6131)在HLA-A*0201I类限制元件背景下以高亲和性识别对应于NY-ESO-1的残基157至165(NY-ESO-1:157-165)的肽SLLMWITQC(SEQ ID NO:25)的突变TCR。该TCR,称为1G4-α95LY,在天然1G4 TCRα链的第三互补性决定区中包含两个氨基酸取代,这赋予了CD8+和CD4+T细胞增强的识别表达NY-ESO-1抗原的HLA-A*0201-阳性靶细胞的能力。最初将TCR IG4-a95LY的整个α链和β链(SEQ ID NO:1和SEQ ID NO:2)或其胞外域(SEQ ID NO:4和SEQ ID NO:5)用于产生天然形式IG4-a95LY TCR的两个抗NY-ESO-1TCR(NT1,NT1b)(SEQ ID NO:9,64)或两个TCR-CAR(IG4-a95LY TCR-CAR)(NT2;基于IG4-a95LYTCR的CAR-CD28Z(aNY-b/a28tm28Z),(SEQ ID NO:10),和NT3;基于IG4-a95LY TCR的CAR-CD28E(aNY-b/a28tm28E)(SEQ ID NO:11)(图1和图2),其在HLA-A*0201I类限制元件背景下以高亲和性识别对应于NY-ESO-1的残基157至165(NY-ESO-1:157-165)的肽SLLMWITQC(SEQID NO:25)。
将上文提及的MGF逆转录病毒骨架用于构建编码天然形式的两个抗NY-ESO-1TCR之一(载体1和载体2;图1A、1B)、十二个基于抗NY-ESO-1TCR的CAR之一(载体113-15;图1A、1B)或抗人间皮素-CAR或GFP的重组病毒载体。
(1)天然形式的抗NY-ESO-1/A2的1G4-a95LY TCR-载体(图1A、图1B、图2A、图2B):
载体1(NT1、NT1a、aNY-TCRa/b)(SEQ ID NO:9和12)
如此前所述(Robbins PF等,J Clin Oncol.2011;29:917-24和Robbins等,JImmunol.2008;180:6116–6131),但在基于MFG的载体中,在逆转录病毒载体骨架中产生编码在HLA-A*0201I类限制元件背景下识别肽SLLMWITQC(SEQ ID NO:25)的TCR的基于MFG的逆转录病毒载体。该TCR,称为1G4-α95:LY,在天然1G4 TCRα链的第三互补性决定区中包含两个氨基酸取代,这赋予了CD8+和CD4+T细胞增强的识别表达NY-ESO-1抗原的HLA-A*0201-阳性靶细胞的能力(Robbins PF等,J Immunol.2008;180:6116–6131)。
在逆转录病毒构建体中表达的1G4α-和β-链在两个基因产物之间含有“自切割”P2A序列(Szymczak等,Nat.Biotechnol.2004;22:589–594)。将NT1的肽序列(aNY-TCRa/b)(SEQ ID NO:9)(图3)设计为由核苷酸序列(SEQ ID NO:12)编码。为了促进在基于MFG的载体的克隆位点中目标基因的分子亚克隆,将XhoI位点(CTCGAG,SEQ ID NO:71)和包含NotI位点(GCGGCCGC,SEQ ID NO:73)的短序列(CAGCCAGCGGCCGC,SEQ ID NO:72)分别插入紧接在编码区中的“ATG”起始密码子的上游和终止密码子“TAA”的下游((SEQ ID NO:12)。
载体2(NT1b;aNY-TCRb/a)
载体(NT2;aNY-TCRb/a)(SEQ ID NO:64和65)的设计与载体1(NT1;aNY-TCRa/b)完全相同,除了编码天然形式TCRα链的DNA片段位于紧邻P2A编码序列3’末端的下游,而β链位于P2A编码序列上游以外(图1、图2和图3)。
(2)与CD28和CD3Z信号传导整合的1G4-a95LY TCR-CAR-载体(NT2、NT3)(图1C和图1D)
载体3(NT2;aNY-b/a28tm28Z)(第二代)
编码两个或更多个在核酸序列上相同或高度同源的分子的重组逆转录病毒可能导致同源重组,结果,可能发生基因组重排,如同源基因的缺失和重复。例如,在质粒DNA和逆转录病毒载体制备过程中,编码基于TCR的CAR的Tm和Cyt域(如CD28TmCyt、CD3ZCyt和CD3ECyt区段)的核酸序列可能导致编码TCR-CAR基因不利的突变和/或缺失。为解决这一问题,可以使用突变程序来抑制同一载体中重复区段之间的同源性驱动的重组。
如此前所述(Govers等,Journal of Immunology,2014,193:5315–5326),将28Z或28E肽序列前面的氨基酸GSPK(SEQ ID NO:69)作为接头(图4和图5),并连接至TCRa(在SPESS的TCR-Ca氨基酸末端)和TCRb(在WGRAD的TCR-Cb氨基酸末端)。
将NT2(载体3;aNY-b/a28tm28Z)(基于1G4 a95LY TCR的CAR)设计为包含28Z信号传导(图1C和图2C)(SEQ ID NO:10)。
为了构建NT2(图2C;SEQ ID NO:10,13),将β链的胞外域(SEQ ID NO:5)和α链链的胞外域(SEQ ID NO:4)通过接头(氨基酸GSPK(SEQ ID NO:69))与人CD28的Tm和Cyt(28TmCyt)(SEQ ID NO:6)和人CD3Z的Cyt(ZCyt)(SEQ ID NO:7)分子连接,其中直接连接到1G4 a95LY TCRα链的胞外域的CD28Z(SEQ ID NO:15)被沉默突变,而没有改变其原始氨基酸序列(mu28muZ)(图6-8)(SEQ ID NO:21),以减少在编码直接连接到β链和α链Ec的C末端的那些序列之间编码28Z的核苷酸序列水平上的同源性。与天然形式的1G4 a95LY TCR(NT1和NT1b)相似,在βCD28Z和αCD28muZ之间也插入了“自切割”P2A序列(SEQ ID NO:3)(图4)。
载体13(NT26;aNY-b/a8h28pectm28Z)(第二代)
以与载体3(NT2)相同的方式设计和制备载体13(NT26,SEQ ID NO:40和62),不同之处是编码人CD8a铰链(CD8h)(SEQ ID NO:30)和部分人CD28胞外域(CD28pec)(SEQ IDNO:26)的相同氨基酸序列的两条DNA序列插入连接至Cb的C末端和CD28Tm的N末端(SEQ IDNO:42,50)的氨基酸GSPK(SEQ ID NO:69)之间和连接至Ca的C末端和CD28Tm的N末端(SEQID NO:43,51)的氨基酸GSPK(SEQ ID NO:69)之间。
(3)与CD28和CD3E信号传导整合的1G4-a95LY TCR-CAR-载体(NT3;aNY-b/a28tm28E)(图1和图2)
载体4(NT3;aNY-b/a28tm28E)(第二代)
28E表达盒在氨基酸序列水平上的设计与此前描述的相同,涵盖了人CD28的Tm和Cyt(Ic)域(GI:338444,aa 153–220,从第一个甲硫氨酸开始编号),随后是人CD3ε的IC域(GI:4502670,aa 153–207)(Tan Van等,JImmunol 2014;193:5315-5326)。28ε表达盒的前面是氨基酸GSPK(SEQ ID NO:69),并连接至Cα区的Ec(TCR-Ca氨基酸以SPESS结尾)下游和TCRb(TCR-Cb氨基酸以WGRAD结尾)。设计了编码NT3(SEQ ID NO:14)的核苷酸序列,其包含紧邻aNY-TCR28E位点编码区上游的Xho I的核苷酸序列以及3主要末端侧翼短片段和Not I位点(图2D、图5和图11)(SEQ ID NO:14)(SEQ ID NO:11),并商业合成(BIO BASIC CANADAINC,Canada)。基于G4 a95LY TCR的CAR包含28E信号传导(NT3;1G4 a95LY TCR-28E)(aNY-b/a28tm28E)(图1D和图2D)。
与aNY-TCR28Z(NT2;载体3)相似,为构建包含28E信号传导的基于IG4 a95LY TCR的CAR(1G4 a95LY TCR-28E;aNY-TCR28E;载体3),将β链和α链的Ec域通过接头(氨基酸GSPK(SEQ ID NO:69))与人CD28的Tm域和Cyt域以及人CD3E的Cyt分子连接,其中直接连接到1G4a95LY TCRα链胞外域的CD28E被沉默突变,而没有改变其原始氨基酸序列(mu28muE)(图6),以减少在编码直接连接到β链和α链Ec的C末端的那些序列之间编码28E的核苷酸序列水平上的同源性。与1G4 a95LY TCR相似,在βCD28E和αmuCD28muE之间也插入了“自切割”P2A序列(SEQ ID NO:3)(图5)。
(4)与4-1BB和CD3Z信号传导整合的1G4-a95LY TCR-CAR-载体(载体5(NT4;aNY-b/a8tmBBZ)和载体6(NT5;aNY-b/a28tmBBZ))
载体5(NT4;aNY-b/a8tmBBZ)(第二代)
以与载体3(NT2)相同的方式设计和制备载体5(SEQ ID NO:32和54),不同之处是编码CD28的Tm和Cyt域的DNA序列被编码人CD8的Tm域(SEQ ID NO:31)(SEQ ID NO:52和53)和人4-1BB的Cyt域(SEQ ID NO:29)(SEQ ID NO:48,49)的DNA序列替代。
载体6(NT5;aNY-b/a28tmBBZ)(第二代)
以与载体5(NT4)相同的方式设计和制备载体6(NT5;aNY-b/a28tmBBZ)(SEQ IDNO:33,55),不同之处是编码CD8的Tm域的DNA序列被编码人CD28的Tm域(SEQ ID NO:29)(SEQ ID NO:48和49)的DNA序列替代。
(5)与CD28和4-1BB和CD3Z信号传导的组合整合的1G4-a95LY TCR-CAR-载体(载体7(NT6)、载体8(NT21)、载体9(NT22)、载体10(NT23)、载体11(NT24)、载体12(NT25)、载体14(NT27))
载体7(NT6;aNY-b/a28tm28BBZ)(第三代)
以与载体3(NT2)相同的方式设计和制备载体7(SEQ ID NO:34,56),不同之处是将编码人4-1BB的Cyt域(SEQ ID NO:29)(SEQ ID NO:48和49)DNA序列分别插入CD28的C末端和CD3Z的N末端的Cyt域之间。
载体11(NT24;aNY-b8tm28Z/a8tmBBZ)(第三代)
以与载体5(NT4;aNY-b/a8tmBBZ)相同的方式设计和制备载体11(SEQ ID NO:38,60),不同之处是编码位于包含TCRβ链的Ec域的第一多肽(即,P2A肽上游)中的CD8Tm的C末端和CD3Zcyt的N末端之间的4-1BB的Cyt域的DNA序列被编码人CD28的Cyt域的DNA序列替代。
载体12(NT25;aNY-b8tm2828Z/a8tmBBBBZ)(第三代)
以与载体11(NT24)相同的方式设计和制备载体12(SEQ ID NO:39和61),不同之处是将编码CD28的Cyt域(CD28Cyt的第2个单元)的另外的DNA序列插入包含抗NY-ESO-1TCRβ链的Ec的第一多肽链中的CD8Tm的C末端和CD29Cyt的N末端之间,以及将编码4-1BB的Cyt域(4-1BBcyt的第2个单元)的另外的DNA序列插入包含抗NY-ESO-1TCRα链的Ec的第二多肽链中的CD8Tm的C末端和4-1BB的N末端之间。
相应地,将与如上文所述相似的分子设计和方法用于设计和制备其他载体,包括载体8(SEQ ID NO:35,57;NT21;aNY-b28tm28Z/a8tmBBZ)、载体9(SEQ ID NO:36,58;NT22;aNY-b28tm28Z/a8tmBB)、载体10(SEQ ID NO:37,59;NT23;aNY-b28tm28Z/a28tmBBZ)、载体14(SEQ ID NO:41,63;NT24;aNY-b8h28pectm28Z/a8tmBB)(表1)。
商业合成(BIO BASIC CANADA INC,Canada)编码14种抗NY-ESO-1 TCR或TCR-CAR(载体1-14;表1)的DNA片段,并且以如此前所述的方法(Yang W等,Int Immunol.2007;19:1083-93)随后亚克隆进入基于MFG的逆转录病毒载体的MCS区的XhoI/NotI位点(图1C)。通过DNA测序验证编码TCR或TCR-CAR的所有载体插入。
实施例2:编码相关目标基因产物的逆转录病毒的产生
用14种载体分别转染Phoenix两性细胞(具有高磷酸钙转染效率的293细胞衍生细胞系)(表1;图1、图2)。根据本领域公知的标准程序,如在Yang W等,Int Immunol.2007;19:1083-93和Beaudoin EL等J Virol Methods 2008;148:253–9中描述的那些,收集转染Phoenix细胞的病毒上清液(含有病毒颗粒),并立即用于感染PG13细胞或其他小鼠细胞,或者在-80℃下保存以供进一步使用。
使用从分别用目标载体感染的Phoenix细胞获得的逆转录病毒上清液感染PG13细胞,一种病毒生产细胞(VPC)系。进行基于FACS的细胞分选以富集相应载体感染的PG13细胞。如此前所述,在使用FITC抗人TCR Vβ13.1抗体(EBIOSCIENCE、AFFYMETRIX和THERMOFISHER SCIENTIFIC)染色后,针对人TCR Vβ13.1链阳性细胞,或NYpep/A2四聚体(四聚体/APC-HLA-A*02:01NY-ESO-1(SLLMWITQC;SEQ ID NO:25)(Fred Hutchinson CancerResearch Center,Seattle,WA 98109,USA))阳性细胞,通过细胞分选富集感染的PG13细胞。按照本领域公知的标准方案,用富集的VPC生产高滴度的逆转录病毒上清液。
在一种情况下,在病毒感染后,TCR+或TCR-CAR+PG13细胞富集的基于FACS的细胞分选之前,先对感染的PG13细胞进行FACS分析。使用从分别用编码3种抗NY-ESO-1TCR-CAR的载体(NT22、NT24和NT25)感染的Phoenix细胞获得的逆转录病毒上清液感染PG13细胞。通过FACS针对TCRVb13.1的表面表达和NYpep/A2四聚体的结合分析感染的PG13细胞系(此前没有通过基于FACS的细胞分选对CAR+细胞进行富集)。对活细胞进行门控以进行分析。图26中显示的强度图分别显示了单个或双重阳性细胞对FITC抗人TCRVb13.1和APC-NYpep/A2四聚体的阳性染色。
实施例3:用逆转录病毒单独转导的活化的人T细胞的产生
以Yang W等,Int Immunol.2007;19:1083-9中所述的方式,将抗CD3抗体活化的人T细胞(ATC)衍生的PBMC分别用以下7种载体的每一种转导:aNY-TCR(NT1,NT1b)和aNY-TCR-CAR(NT2、NT3、NT4和NT5、NT6)(表1)。转导后7至10天,通过使用FITC-抗人TCR Vβ13.1和四聚体/APC-HLA-A*02:01NY-ESO-1(SLLMWITQC,SEQ ID NO:5)(Fred Hutchinson CancerResearch Center,Seattle,WA)的组合染色进行流式细胞术分析。结果如图27中所示。
如图中所示,与天然形式(NT1和NT1b)相比,两个第二代抗NYESO-1/A2TCR-CAR(NT2和NT3)针对人TCRVb13.1和NYpep/A2四聚体结合TCR两者在转导的ATC(包括CD4+和CD8+细胞)细胞上均以显著更高水平表达。约3.9%的未转导细胞(Untd)针对TCRVb13.1染色也是阳性的,但是这些细胞未被APC-NYpep/A2四聚体显著染色。
发现19.4-51.2%之间的活化的T细胞与四聚体/APC-HLA-A*02:01NY-ESO-1结合。基于FITC抗人TCR Vβ13.1抗体(EBIOSCIENCE/AFFYMETRIX,CA)染色测量在其细胞表面上TCR Vβ13.1的表达,以及基于使用四聚体/PE-HLA-A*02:01NY-ESO-1的阳性四聚体染色以及随后的FACS分析测量在细胞表面上功能性1G4 a95LY TCR Ec域的校正对表达的活化T细胞的相似百分比。进一步的分析表明,感染的活化人T细胞的CAR+CD3+细胞的大约一半是CD4+(52%)和CD8+(48%)。
实施例4:使用抗NY-ESO-1TCR或两种基于TCR的CAR之一转导的人ATC的抗原特异性介导的细胞因子IL-2分泌
在本实施例中,通过在不存在和存在各种量的特异性抗原肽的情况下,检测在其中T细胞与HLA-A0201+靶细胞共培养的培养基中IL-2的分泌,考察活化的人T细胞诱导细胞因子分泌的活性。
简言之,在不存在或存在各种浓度的肽NY-ESO-1(SLLMWITQC,SEQ ID NO:25,10nM、100nM和2uM)的情况下,将0.5x105个HLA-A0201+靶细胞(T2细胞,174x CEM.T2,ATCCCRL-1992TM)在培养基中在37℃下孵育3小时。将细胞洗涤两次以除去未结合的肽,然后与1.5x105个活化的T细胞在U底96孔培养板每孔的200ul培养基(完全RPMI 1640+10%FCS+Pen/Strep)(R-10)中共培养24小时,所述T细胞分别使用载体NT1(NT1a)、NT1b、NT2和NT3转导(图2)。
收集培养上清液,并通过ELISA检测试剂盒(EBIOSCIENCES)测量IL-2的浓度。所得到的IL-2分泌数据表明,在与NY-ESO-1肽脉冲T2细胞,而非未使用NY-ESO-1肽脉冲或使用阴性对照肽脉冲的T2细胞接合后,分别使用所有三种抗NY-ESO-1载体NT1(NT1a)、NT1b、NT2和NT3(图2)转导的活化的T细胞特异性地分泌IL-2。
发现用NT2转导的T细胞在这三种中分泌最高量的IL-2,而用NT1转导的T细胞在脉冲T2细胞的相同肽浓度下产生最低量的IL-2。这些结果表明,在转导的TCR或TCR-CAR介导的T细胞活化中,两种TCR-CAR(NT2和NT3)优于NT1(图2A)。
实施例5:使用抗NY ESO-1TCR或两种基于TCR的CAR之一转导的ATC的特异性肿瘤细胞杀伤作用
在本实施例中,使用Saos-2细胞(HTB-85TM)进行测定,以考察使用抗NY-ESO-1TCR或基于TCR的载体NT1、NT1b、NT2和NT3(图2)转导的活化的人T细胞(ATC)的肿瘤细胞杀伤活性。Saos-2细胞系(一种人骨肉瘤细胞系)是NY-ESO-1+和HLA-A0201+的。
简言之,将1x103个Saos-2细胞在96孔平底组织培养板上预先铺板12小时。然后,将1x104个未转导或转导的人T细胞加入每孔中。在补充了90IU rec hu IL-2/ml的完全RPMI 1640+10%FCS+Pen/Strep培养基(R10)中的这些T细胞是未转导或已分别使用三种载体NT1、NT2和NT3转导的。将细胞培养至多5天(120hr)(图28)。存活的肿瘤靶细胞附着在孔的底部,其远大于T细胞并且与T细胞截然不同,并且每天进行观察和分级。
将T细胞的杀伤活性分级为:“++++”,75-100%杀伤;“++++”,75-100%杀伤;“+++”,50-75%杀伤;“++”,25-50%杀伤;“+”,0-25%杀伤,通过对含有T细胞的孔的存活肿瘤细胞数与不含T细胞的孔的存活肿瘤细胞数进行比较。在添加T细胞后第5天(120hr),在显微镜下拍摄孔的代表性视图的照片。见图28。
如图中所示,表达aNY-TCRa/b的ATC(NT1(NT1a)和NT1b)显示出相似的,最高的肿瘤杀伤活性。表达NT2的ATC显示出略低的杀伤活性,而具有NT3的那些显示出相对较低的杀伤活性。未转导的ATC没有显示出明显的杀伤作用。这些结果表明,分别使用四种载体转导的人T细胞能够特异性杀伤Saos-2肿瘤细胞。
实施例6:抗原特异性介导的细胞因子IL-2和INF-γ的分泌
在本实施例中,进行测定以考察使用两种抗NY ESO-1TCR(NT1(NT1a)和NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的ATC的细胞因子分泌。
以上文所述的方法制备使用NT1a、NT1b、NT2或NT3转导的ATC。如上文所述,使用未转染的T细胞(“untd”),将基于通过FACS的APC-NYpep/A2四聚体染色(针对NT1a、NT1b、NT2和NT3)的NYpep/A2阳性T细胞的百分比调整为20%。然后,将0.5x106个靶细胞(Saos-2或对照AspC1)或仅培养基(RPMI-1640+10%FCS+Pen/Strep)(R10)(未添加任何IL-2)与使用NT1a、NT1b、NT2或NT3转导的2x106个ATC在24孔平底组织培养板中以R10的总体积1.5mL(未添加任何IL-2)培养24小时。用来自BIOLEGENDS的试剂盒(目录号431804和430101)通过ELISA测量细胞上清液中IL-2或INF-γ的浓度。结果如图29中所示。如图29A中所示,使用NT2转导的T细胞分泌最高量的IL-2(约230pg/ml),而使用NT3的那些分泌中等量的IL-2,使用两种天然形式抗NY-ESO-1/A2 TCR(NT1和NT1b)的每一种的那些分泌最低量。
实施例7:使用NT1、NT1b、NT2和NT3转导的ATC的NYpep/A2+细胞的百分比变化
以上文所述的方式制备使用两种抗NY ESO-1TCR(NT1和NT1b)之一或两种第二代基于TCR的CAR(NT2和NT3)之一转导的ATC。如上文所述,使用untd的T细胞,将基于通过FACS的APC-NYpep/A2四聚体染色(针对NT1a、NT1b、NT2和NT3)的NYpep/A2阳性T细胞的百分比调整为20%。
在第1天,将1x106个靶细胞(Saos-2)与4x106个使用NT1a、NT1b、NT2或NT3转导的ATC在12孔平底组织培养瓶中在总体积4ml含90IU IL-2/ml的R10中共培养48小时。在第3天,将细胞转移至新的24孔板。在第6天,将来自每个孔的细胞分成两个孔,所述孔中具有含180IU IL-2/ml的R10,总体积为4ml/孔。在第10天,收获细胞并通过FACS分析NYpep/A2四聚体的结合。结果如图30中所示。如图中所示,对活细胞进行门控以进行分析,并显示阳性细胞的百分比。发现在NT2中NYpep/A2阳性的百分比最显著增加(约32%),而在NT3中观察到中度增加(约25%)和在NT1a和NT1b组中观察到降低(约17-19%)。
实施例8:第二代TCR-CAR和第三代TCR-CAR的分析
在本实施例中,在转导的活化人T细胞上进行测定以考察比较天然形式的抗NY-ESO-1TCR以及第二代或第三代TCR-CAR。
首先,考察TCR或TCR-CAR在转导的活化人T细胞上的表面表达。简言之,抗CD3抗体活化的人T细胞是未转导的(Untd),或者使用含有病毒的上清液转导的,所述上清液来自如上文所述的此前分选的抗NY-ESO-1TCR+(NT1(也称为NT1a))或抗NY-ESO-1TCR-CAR+((NT2;第二代)、NT4(载体5;第二代)、NT5(载体6;第二代)和NT24(载体11;第三代))细胞系。转导后7至10天,通过FACS针对TCRVb13.1的表面表达和NYpep/A2四聚体的结合对转导的T细胞进行分析。对活细胞进行门控以进行分析。结果如图31A和图31B所示。如图中所示,如通过TCRVb13.1和NYpep/A2四聚体染色利用FACS所确定的,使用三种第二代抗NY-ESO-1TCR-CAR(NT2、NT4和NT5)的每一种转导的T细胞表达最多的和相似最高水平的TCRVb13.1和NYpep/A2四聚体-结合TCR或TCR-CAR。使用第三代TCR-CAR NT24的那些显示出相对较低的双重TCRVb13.1和NYpep/A2四聚体结合+细胞,而使用天然形式TCR(NT1)的那些显示出最低的双重TCRVb13.1和NYpep/A2四聚体结合+细胞百分比。
其次,在与靶细胞接合后,进行测定以考察使用抗NY ESO-1TCR或四种TCR-CAR之一转导的人ATC的靶细胞杀伤能力。简言之,如上所述制备使用NT1(也称为NT1a)、NT2、NT4、NT5或NT24转导的ATC。在存在细胞因子IL-2的情况下,在其中转导的人T细胞(效应物)与Saos-2肿瘤细胞(靶点)共培养的体外杀伤测定中进行针对这些活化的人T细胞的抗原特异性靶细胞杀伤。在实验中,使用untd T细胞,将分别使用NT1a、NT2、NT4、NT5或NT24转导的人T细胞调整为15%NYpep/A+,然后在存在细胞因子IL-2的情况下分别以E/T比率1:2和1:20与Saos-2肿瘤细胞(靶点)共培养12hr。在孵育结束时,使用Cayman 7-AAD/CFSE细胞介导的细胞毒性测定试剂盒测量T细胞介导的细胞毒性。结果如图32中所示,其中数据以来自每组中三个样品的平均值±S.D.表示。如图32中所示,在这两个E/T比率下(1:2和1:20),使用NT1a之一和三个基于TCR的CAR(NT4、NT5和NT24)之一转导的T细胞显示出最高的和相似的靶细胞杀伤能力,所述基于TCR的CAR包含来源于4-1BB的TCR信号2。使用NT2(第二代的TCR-CRA,仅包含来源于CD28的TCR信号2)的那些显示出略低的杀伤能力。使用未转导的细胞(Untd)未见显著的靶细胞杀伤作用。
再次,考察了与靶细胞接合后使用TCR或基于TCR的CAR转导的人ATC的细胞因子分泌情况。如上文所述制备使用NT1a、NT2、NT4、NT5或NT24转导的ATC。使用untd T细胞,将转导的T细胞再调整为15%NYpep/A+。然后,将0.5x106个靶Saos-2细胞或仅培养基(R10)与2x106个转导的ATC或未转导的人T细胞(Untd)在24孔平底组织培养板中以总体积1.5ml的R10(未添加任何IL-2)共培养24小时。如上文所述通过ELISA测量细胞上清液中细胞因子的浓度。结果如图33A和图33B中所示,其中数据以来自每组中三个样品的平均值±S.D.表示。如图33A中所示,使用NT2或NT24(均包含来源于人CD28的1个(NT24)或2个(NT2)TCR信号传导2)的那些分泌最高量的IL-2(分别为约200pg/ml和215pg/ml)。使用NT4或NT5(具有来自4-1BB的TCR信号2)的那些产生中等的量(分别为约170pg/ml和165pg/ml)。使用天然形式抗NY-ESO-1/A2 TCR(NT1)的那些分泌最低量。对于IFN-γ,如图33B所示,在与靶Saos-2细胞接合后,而非不存在靶细胞(R10对照)的情况下,使用NT1(也称为NT1a)、NT2、NT4、NT5和NT24转导的ATC分别相似水平的IFN-γ。在存在或不存在靶细胞的情况下,阴性对照细胞Untd未产生显著的量的IL-2或IFN-r。
最后,进行测定以考察与靶细胞接合后使用抗NY ESO-1TCR或四种基于TCR的CAR之一转导的NYpep/A2+ATC的百分比和扩增的变化。简言之,在第1天,使用untd细胞,将使用NT1a、NT2、NT4、NT5或NT24转导的ATC调整为15%NYpep/A+。然后,将1x106个靶Saos-2细胞与6x106个ATC在12孔平底组织培养板中以总体积5ml的含90IU IL-2/ml的R10共培养48小时,所述ATC此前已调整为15%NYpep/A+细胞并且由0.9x106个NYpep/A+细胞组成。在第3天时,将所有细胞转移至新的12孔板。在第6天(共培养开始后6天),将每个孔的细胞分成两个新孔,所述孔中具有含180IU IL-2/ml的R10,总体积为5ml/孔。在第10天,收集悬浮细胞,使用台盼蓝染色在显微镜检查下计数,并通过FACS对FITC-抗人CD3抗体和APC-NYpep/A2四聚体进行分析。发现超过95-98%的活细胞是CD3+的(数据未显示)。对活细胞和CD3+细胞进行门控以进行分析。结果如图3A和图3B中所示。根据以下公式计算NYpep/A2+ATC的总数:
如图34A中所示的活细胞总数(通过台盼蓝染色)占NYpep/A2+细胞的%/NYpep/A2+ATC总数。
如图34A中所示,在所有四个TCR-CAR(NT2、NT4、NT5和NT24)中,NYpep/A2阳性细胞的百分比从15%显著增至相似水平,范围从25.3%至29.2%;分别为28.4%、27.1%、25.3%和29.2%。NT1a的增加略增加至19.1%。如图34B中所示,针对NT2、NT4、NT5和NT24的增加倍数分别为3.2、3.1、2.9和3.5,而针对NT1为1.2。
实施例9:体内抗肿瘤活性
在本实施例中,将异种移植小鼠模型用于考察使用本文所述的载体转导的T细胞的体内抗肿瘤活性。
简言之,在效应T细胞的过继细胞转移前5天(第-5天),在100ul PBS中(以例如3x106个细胞/小鼠),将Saos-2细胞皮下注射至SCID雌性小鼠的后胁部。如上述图31中所述制备分别使用编码NT1a、NT2、NT4和NT24的逆转录病毒转导的活化的人T细胞或未转导的人T细胞(Untd)。
在第0天,将小鼠分成五组,每组六只小鼠。使用未转导的T细胞将使用NT1a、NT2、NT4或NT24转导的ATC调整至15%NYpep/A+。然后,以1x107个T细胞/100ul PBS/小鼠的剂量,将细胞(含有CD4+和CD8+T细胞)通过尾静脉静脉内施用。
监测小鼠的肿瘤生长。使用卡尺测量肿瘤尺寸,并使用公式(肿瘤测量结果(mm3)=长度x宽度x高度x0.5236)计算肿瘤体积。由于肿瘤尺寸过大而处死的小鼠或发现死亡的小鼠被排除在肿瘤尺寸数据的收集和分析之外。结果如图35所示。如图中所示,在ACT施用后第54天,仅NT24组全部6只小鼠(100%)仍存活。而相反的是,在NT1、NT2、NT4和Untd组中分别有0、4、4和0只小鼠存活。此外,在ATC后第62天实验结束时,尽管分别在NT1、NT2、NT4和Untd组中6只小鼠均未存活,但是在NT24组的6只小鼠中有3只存活(50%)。
这些体内数据表明,第三代抗NY-ESO-1/A2 TCR-CAR NT24的效力明显高于仅具有来自人CD28(NT2)或仅具有来自4-1BB(NT4)的TCR信号2的第二代其对应部分,甚至比天然形式NT1(NT1a)更高效。数据还表明这两种第二代抗NY-ESO-1TCR-CAR均显示出比天然形式NT1(NT1a)更优异的抗肿瘤活性。
应当将前述实施例和具体实施方式用作说明,而不是限制由权利要求书限定的本发明。如将容易理解的是,在不脱离权利要求中所阐述的本发明的情况下,可以利用上述特征的多种变化和组合。不认为这样的变形脱离了本发明的范围,并且所有这样的变形旨在包括在所附权利要求的范围内。本文中引用的所有参考文献通过引用整体并入本文。
序列表
<110> Yang, Wen
<120> 用于过继免疫疗法的组合物和方法
<130> 180423.00200
<150> 62/877,331
<151> 2019-07-23
<150> 63/044,059
<151> 2020-06-25
<160> 73
<170> PatentIn version 3.5
<210> 1
<211> 275
<212> PRT
<213> 智人
<400> 1
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
115 120 125
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser Arg
275
<210> 2
<211> 311
<212> PRT
<213> 智人
<400> 2
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 3
<211> 19
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 3
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1 5 10 15
Pro Gly Pro
<210> 4
<211> 227
<212> PRT
<213> 智人
<400> 4
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
115 120 125
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser
225
<210> 5
<211> 262
<212> PRT
<213> 智人
<400> 5
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp
260
<210> 6
<211> 68
<212> PRT
<213> 智人
<400> 6
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
20 25 30
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
35 40 45
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
50 55 60
Ala Tyr Arg Ser
65
<210> 7
<211> 112
<212> PRT
<213> 智人
<400> 7
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 8
<211> 55
<212> PRT
<213> 智人
<400> 8
Lys Asn Arg Lys Ala Lys Ala Lys Pro Val Thr Arg Gly Ala Gly Ala
1 5 10 15
Gly Gly Arg Gln Arg Gly Gln Asn Lys Glu Arg Pro Pro Pro Val Pro
20 25 30
Asn Pro Asp Tyr Glu Pro Ile Arg Lys Gly Gln Arg Asp Leu Tyr Ser
35 40 45
Gly Leu Asn Gln Arg Arg Ile
50 55
<210> 9
<211> 612
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 9
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
115 120 125
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser Arg Ala Lys Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu
275 280 285
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Ile
290 295 300
Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala Gly Pro Val
305 310 315 320
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
325 330 335
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Glu Tyr Met
340 345 350
Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
355 360 365
Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro Asn Gly Tyr
370 375 380
Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg Leu Leu Ser
385 390 395 400
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Val
405 410 415
Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu Thr Val
420 425 430
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
435 440 445
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
450 455 460
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
465 470 475 480
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
485 490 495
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu Ser Ser Arg
500 505 510
Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn His Phe Arg
515 520 525
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
530 535 540
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
545 550 555 560
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
565 570 575
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
580 585 590
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
595 600 605
Asp Ser Arg Gly
610
<210> 10
<211> 883
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 10
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
370 375 380
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
385 390 395 400
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
405 410 415
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
420 425 430
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys
435 440 445
Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
450 455 460
Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu
465 470 475 480
Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr
485 490 495
Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu
500 505 510
Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg
515 520 525
Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser
530 535 540
Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser
545 550 555 560
Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser
565 570 575
Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile
580 585 590
Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln
595 600 605
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
610 615 620
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
625 630 635 640
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
645 650 655
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
660 665 670
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
675 680 685
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Phe
690 695 700
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
705 710 715 720
Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg
725 730 735
Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro
740 745 750
Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala
755 760 765
Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
770 775 780
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
785 790 795 800
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
805 810 815
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
820 825 830
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
835 840 845
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
850 855 860
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
865 870 875 880
Pro Pro Arg
<210> 11
<211> 769
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 11
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Asn
325 330 335
Arg Lys Ala Lys Ala Lys Pro Val Thr Arg Gly Ala Gly Ala Gly Gly
340 345 350
Arg Gln Arg Gly Gln Asn Lys Glu Arg Pro Pro Pro Val Pro Asn Pro
355 360 365
Asp Tyr Glu Pro Ile Arg Lys Gly Gln Arg Asp Leu Tyr Ser Gly Leu
370 375 380
Asn Gln Arg Arg Ile Ala Lys Arg Ser Gly Ser Gly Ala Thr Asn Phe
385 390 395 400
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
405 410 415
Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val
420 425 430
Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro
435 440 445
Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile
450 455 460
Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser
465 470 475 480
Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu
485 490 495
Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala
500 505 510
Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro
515 520 525
Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser Leu
530 535 540
Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
545 550 555 560
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
565 570 575
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
580 585 590
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
595 600 605
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
610 615 620
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
625 630 635 640
Ser Ser Gly Ser Pro Lys Phe Trp Val Leu Val Val Val Gly Gly Val
645 650 655
Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp
660 665 670
Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met
675 680 685
Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala
690 695 700
Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Asn Arg Lys Ala Lys
705 710 715 720
Ala Lys Pro Val Thr Arg Gly Ala Gly Ala Gly Gly Arg Gln Arg Gly
725 730 735
Gln Asn Lys Glu Arg Pro Pro Pro Val Pro Asn Pro Asp Tyr Glu Pro
740 745 750
Ile Arg Lys Gly Gln Arg Asp Leu Tyr Ser Gly Leu Asn Gln Arg Arg
755 760 765
Ile
<210> 12
<211> 1839
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 12
atggagaccc tcttgggcct gcttatcctt tggctgcagc tgcaatgggt gagcagcaaa 60
caggaggtga cgcagattcc tgcagctctg agtgtcccag aaggagaaaa cttggttctc 120
aactgcagtt tcactgatag cgctatttac aacctccagt ggtttaggca ggaccctggg 180
aaaggtctca catctctgtt gcttattcag tcaagtcaga gagagcaaac aagtggaaga 240
cttaatgcct cgctggataa atcatcagga cgtagtactt tatacattgc agcttctcag 300
cctggtgact cagccaccta cctctgtgct gtgaggcccc tgtacggagg aagctacata 360
cctacatttg gaagaggaac cagccttatt gttcatccgt atatccagaa ccctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctgtggtcca gccgggccaa gcggtctggg 840
tctggggcca ccaacttcag cctgctgaag caggccggcg acgtggagga gaaccccggc 900
cccatgagca tcggcctcct gtgctgtgca gccttgtctc tcctgtgggc aggtccagtg 960
aatgctggtg tcactcagac cccaaaattc caggtcctga agacaggaca gagcatgaca 1020
ctgcagtgtg cccaggatat gaaccatgaa tacatgtcct ggtatcgaca agacccaggc 1080
atggggctga ggctgattca ttactcagtt ggtgctggta tcactgacca aggagaagtc 1140
cccaatggct acaatgtctc cagatcaacc acagaggatt tcccgctcag gctgctgtcg 1200
gctgctccct cccagacatc tgtgtacttc tgtgccagca gttacgtcgg gaacaccggg 1260
gagctgtttt ttggagaagg ctctaggctg accgtactgg aggacctgaa aaacgtgttc 1320
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 1380
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 1440
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 1500
gccctcaatg actccagata cgctctgagc agccgcctga gggtctcggc caccttctgg 1560
caggaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 1620
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 1680
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 1740
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 1800
ctgatggcta tggtcaagag aaaggattcc agaggctaa 1839
<210> 13
<211> 2652
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 13
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtcgggtcaa attttcacgc 1020
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1080
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1140
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1200
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1260
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1320
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1380
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1440
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1500
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1560
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1620
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1680
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1740
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1800
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1860
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 1920
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 1980
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2040
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2100
tccccaaaat tttgggtgct ggtggtggtt ggtggagtcc tggcttgcta tagcttgcta 2160
gtaacagtgg cctttattat tttctgggtg aggagtaaga ggagcaggct cctgcacagt 2220
gactacatga acatgactcc ccgccgcccc gggcccaccc gcaagcatta ccagccctat 2280
gccccaccac gcgacttcgc agcctatcgc tccagagtga agttcagcag gagcgcagac 2340
gcccccgcgt accagcaggg ccagaaccag ctctataacg agctcaatct aggacgaaga 2400
gaggagtacg atgttttgga caagagacgt ggccgggacc ctgagatggg gggaaagccg 2460
agaaggaaga accctcagga aggcctgtac aatgaactgc agaaagataa gatggcggag 2520
gcctacagtg agattgggat gaaaggcgag cgccggaggg gcaaggggca cgatggcctt 2580
taccagggtc tcagtacagc caccaaggac acctacgacg cccttcacat gcaggccctg 2640
ccccctcgct aa 2652
<210> 14
<211> 2310
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 14
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtaaaaaccg caaagctaaa 1020
gctaaacccg tcactagggg ggccggagca ggagggcgcc agcgcggtca gaataaagaa 1080
cgccctcctc ccgtccctaa tcctgattac gaaccgatta gaaaggggca aagagatctc 1140
tacagcggac tcaaccaacg gagaattgcc aagcggtctg ggtctggggc caccaacttc 1200
agcctgctga agcaggccgg cgacgtggag gagaaccccg gccccatgga gaccctcttg 1260
ggcctgctta tcctttggct gcagctgcaa tgggtgagca gcaaacagga ggtgacgcag 1320
attcctgcag ctctgagtgt cccagaagga gaaaacttgg ttctcaactg cagtttcact 1380
gatagcgcta tttacaacct ccagtggttt aggcaggacc ctgggaaagg tctcacatct 1440
ctgttgctta ttcagtcaag tcagagagag caaacaagtg gaagacttaa tgcctcgctg 1500
gataaatcat caggacgtag tactttatac attgcagctt ctcagcctgg tgactcagcc 1560
acctacctct gtgctgtgag gcccctgtac ggaggaagct acatacctac atttggaaga 1620
ggaaccagcc ttattgttca tccgtatatc cagaaccctg accctgccgt gtaccagctg 1680
agagactcta aatccagtga caagtctgtc tgcctattca ccgattttga ttctcaaaca 1740
aatgtgtcac aaagtaagga ttctgatgtg tatatcacag acaaaactgt gctagacatg 1800
aggtctatgg acttcaagag caacagtgct gtggcctgga gcaacaaatc tgactttgca 1860
tgtgcaaacg ccttcaacaa cagcattatt ccagaagaca ccttcttccc cagcccagaa 1920
agttccggct ccccaaaatt ttgggtgctg gtggtggttg gtggagtcct ggcttgctat 1980
agcttgctag taacagtggc ctttattatt ttctgggtga ggagtaagag gagcaggctc 2040
ctgcacagtg actacatgaa catgactccc cgccgccccg ggcccacccg caagcattac 2100
cagccctatg ccccaccacg cgacttcgca gcctatcgct ccaagaatag aaaggccaag 2160
gccaagcctg tgacacgagg agcgggtgct ggcggcaggc aaaggggaca aaacaaggag 2220
aggccaccac ctgttcccaa cccagactat gagcccatcc gcaaaggcca gcgggacctg 2280
tattctggcc tgaatcagag acgcatctaa 2310
<210> 15
<211> 141
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 15
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
1 5 10 15
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys
20 25 30
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
35 40 45
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
50 55 60
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
65 70 75 80
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
85 90 95
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
100 105 110
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
115 120 125
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
130 135 140
<210> 16
<211> 540
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 16
ttttgggtgc tggtggtggt tggtggagtc ctggcttgct atagcttgct agtaacagtg 60
gcctttatta ttttctgggt gaggagtaag aggagcaggc tcctgcacag tgactacatg 120
aacatgactc cccgccgccc cgggcccacc cgcaagcatt accagcccta tgccccacca 180
cgcgacttcg cagcctatcg ctccagagtg aagttcagca ggagcgcaga cgcccccgcg 240
taccagcagg gccagaacca gctctataac gagctcaatc taggacgaag agaggagtac 300
gatgttttgg acaagagacg tggccgggac cctgagatgg ggggaaagcc gagaaggaag 360
aaccctcagg aaggcctgta caatgaactg cagaaagata agatggcgga ggcctacagt 420
gagattggga tgaaaggcga gcgccggagg ggcaaggggc acgatggcct ttaccagggt 480
ctcagtacag ccaccaagga cacctacgac gcccttcaca tgcaggccct gccccctcgc 540
<210> 17
<211> 204
<212> DNA
<213> 智人
<400> 17
ttttgggtgc tggtggtggt tggtggagtc ctggcttgct atagcttgct agtaacagtg 60
gcctttatta ttttctgggt gaggagtaag aggagcaggc tcctgcacag tgactacatg 120
aacatgactc cccgccgccc cgggcccacc cgcaagcatt accagcccta tgccccacca 180
cgcgacttcg cagcctatcg ctcc 204
<210> 18
<211> 204
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 18
ttctgggttc tcgtcgtcgt gggaggtgtg ttagcatgtt actctctctt ggttactgtc 60
gctttcataa tcttttgggt ccgctcaaaa cgctctcgct tgttacattc cgattatatg 120
aatatgacac ctaggagacc tggcccgact aggaaacact atcaacctta cgcacctccc 180
agagattttg ctgcttacag gagt 204
<210> 19
<211> 336
<212> DNA
<213> 智人
<400> 19
agagtgaagt tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc 60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc 120
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 180
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 240
cggaggggca aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc 300
tacgacgccc ttcacatgca ggccctgccc cctcgc 336
<210> 20
<211> 336
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 20
cgggtcaaat tttcacgctc cgctgatgct cctgcctatc aacaagggca aaatcaattg 60
tacaatgaat tgaacttggg tagaagggaa gaatatgacg tgctcgataa acggaggggg 120
agagatccag aaatgggcgg taaaccacgg cgcaaaaatc cacaagaggg attgtataac 180
gagctccaaa aggacaaaat ggcagaagct tattcagaaa taggaatgaa gggggaaagg 240
agacgaggta aaggtcatga cggattgtat caaggattgt caaccgctac taaagataca 300
tatgatgctt tgcatatgca agctttgcct cccaga 336
<210> 21
<211> 540
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 21
ttctgggttc tcgtcgtcgt gggaggtgtg ttagcatgtt actctctctt ggttactgtc 60
gctttcataa tcttttgggt ccgctcaaaa cgctctcgct tgttacattc cgattatatg 120
aatatgacac ctaggagacc tggcccgact aggaaacact atcaacctta cgcacctccc 180
agagattttg ctgcttacag gagtcgggtc aaattttcac gctccgctga tgctcctgcc 240
tatcaacaag ggcaaaatca attgtacaat gaattgaact tgggtagaag ggaagaatat 300
gacgtgctcg ataaacggag ggggagagat ccagaaatgg gcggtaaacc acggcgcaaa 360
aatccacaag agggattgta taacgagctc caaaaggaca aaatggcaga agcttattca 420
gaaataggaa tgaaggggga aaggagacga ggtaaaggtc atgacggatt gtatcaagga 480
ttgtcaaccg ctactaaaga tacatatgat gctttgcata tgcaagcttt gcctcccaga 540
<210> 22
<211> 165
<212> DNA
<213> 智人
<400> 22
aagaatagaa aggccaaggc caagcctgtg acacgaggag cgggtgctgg cggcaggcaa 60
aggggacaaa acaaggagag gccaccacct gttcccaacc cagactatga gcccatccgg 120
aaaggccagc gggacctgta ttctggcctg aatcagagac gcatc 165
<210> 23
<211> 165
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 23
aagaatagaa aggccaaggc caagcctgtg acacgaggag cgggtgctgg cggcaggcaa 60
aggggacaaa acaaggagag gccaccacct gttcccaacc cagactatga gcccatccgc 120
aaaggccagc gggacctgta ttctggcctg aatcagagac gcatc 165
<210> 24
<211> 165
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 24
aaaaaccgca aagctaaagc taaacccgtc actagggggg ccggagcagg agggcgccag 60
cgcggtcaga ataaagaacg ccctcctccc gtccctaatc ctgattacga accgattaga 120
aaggggcaaa gagatctcta cagcggactc aaccaacgga gaatt 165
<210> 25
<211> 9
<212> PRT
<213> 智人
<400> 25
Ser Leu Leu Met Trp Ile Thr Gln Cys
1 5
<210> 26
<211> 40
<212> PRT
<213> 智人
<400> 26
Lys Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser
1 5 10 15
Asn Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro
20 25 30
Leu Phe Pro Gly Pro Ser Lys Pro
35 40
<210> 27
<211> 27
<212> PRT
<213> 智人
<400> 27
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 28
<211> 41
<212> PRT
<213> 智人
<400> 28
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 29
<211> 42
<212> PRT
<213> 智人
<400> 29
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 30
<211> 46
<212> PRT
<213> 智人
<400> 30
Ala Lys Pro Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
1 5 10 15
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
20 25 30
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
35 40 45
<210> 31
<211> 26
<212> PRT
<213> 智人
<400> 31
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
1 5 10 15
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
20 25
<210> 32
<211> 883
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 32
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Cys Asp Ile Tyr Ile Trp
260 265 270
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
275 280 285
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
290 295 300
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
305 310 315 320
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
370 375 380
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
385 390 395 400
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
405 410 415
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
420 425 430
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys
435 440 445
Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
450 455 460
Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu
465 470 475 480
Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr
485 490 495
Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu
500 505 510
Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg
515 520 525
Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser
530 535 540
Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser
545 550 555 560
Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser
565 570 575
Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile
580 585 590
Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln
595 600 605
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
610 615 620
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
625 630 635 640
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
645 650 655
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
660 665 670
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
675 680 685
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Cys
690 695 700
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
705 710 715 720
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
725 730 735
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
740 745 750
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
755 760 765
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
770 775 780
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
785 790 795 800
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
805 810 815
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
820 825 830
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
835 840 845
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
850 855 860
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
865 870 875 880
Pro Pro Arg
<210> 33
<211> 885
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 33
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
290 295 300
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
305 310 315 320
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
325 330 335
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
340 345 350
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
355 360 365
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
370 375 380
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
385 390 395 400
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
405 410 415
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
420 425 430
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala
435 440 445
Lys Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala
450 455 460
Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu
465 470 475 480
Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val
485 490 495
Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val
500 505 510
Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe
515 520 525
Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser
530 535 540
Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys
545 550 555 560
Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp
565 570 575
Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr
580 585 590
Ile Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile
595 600 605
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
610 615 620
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
625 630 635 640
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
645 650 655
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
660 665 670
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
675 680 685
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys
690 695 700
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
705 710 715 720
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Lys Arg Gly Arg Lys
725 730 735
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
740 745 750
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
755 760 765
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
770 775 780
Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
785 790 795 800
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
805 810 815
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
820 825 830
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
835 840 845
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
850 855 860
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
865 870 875 880
Ala Leu Pro Pro Arg
885
<210> 34
<211> 967
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 34
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg
325 330 335
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
340 345 350
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
355 360 365
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
370 375 380
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
385 390 395 400
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
405 410 415
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
420 425 430
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
435 440 445
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
450 455 460
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
465 470 475 480
His Met Gln Ala Leu Pro Pro Arg Ala Lys Arg Ser Gly Ser Gly Ala
485 490 495
Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
500 505 510
Gly Pro Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu
515 520 525
Gln Trp Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu
530 535 540
Ser Val Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp
545 550 555 560
Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly
565 570 575
Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser
580 585 590
Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu
595 600 605
Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala
610 615 620
Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly
625 630 635 640
Thr Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val
645 650 655
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
660 665 670
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
675 680 685
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
690 695 700
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
705 710 715 720
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
725 730 735
Ser Pro Glu Ser Ser Gly Ser Pro Lys Phe Trp Val Leu Val Val Val
740 745 750
Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile
755 760 765
Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
770 775 780
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
785 790 795 800
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly
805 810 815
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
820 825 830
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
835 840 845
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
850 855 860
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
865 870 875 880
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
885 890 895
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
900 905 910
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
915 920 925
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
930 935 940
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
945 950 955 960
Met Gln Ala Leu Pro Pro Arg
965
<210> 35
<211> 883
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 35
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
370 375 380
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
385 390 395 400
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
405 410 415
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
420 425 430
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys
435 440 445
Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
450 455 460
Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu
465 470 475 480
Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr
485 490 495
Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu
500 505 510
Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg
515 520 525
Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser
530 535 540
Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser
545 550 555 560
Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser
565 570 575
Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile
580 585 590
Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln
595 600 605
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
610 615 620
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
625 630 635 640
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
645 650 655
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
660 665 670
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
675 680 685
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Cys
690 695 700
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
705 710 715 720
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
725 730 735
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
740 745 750
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
755 760 765
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
770 775 780
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
785 790 795 800
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
805 810 815
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
820 825 830
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
835 840 845
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
850 855 860
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
865 870 875 880
Pro Pro Arg
<210> 36
<211> 771
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 36
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
370 375 380
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
385 390 395 400
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
405 410 415
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
420 425 430
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys
435 440 445
Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
450 455 460
Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu
465 470 475 480
Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr
485 490 495
Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu
500 505 510
Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg
515 520 525
Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser
530 535 540
Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser
545 550 555 560
Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser
565 570 575
Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile
580 585 590
Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln
595 600 605
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
610 615 620
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
625 630 635 640
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
645 650 655
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
660 665 670
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
675 680 685
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Cys
690 695 700
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
705 710 715 720
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
725 730 735
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
740 745 750
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
755 760 765
Cys Glu Leu
770
<210> 37
<211> 884
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 37
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Phe Trp Val Leu Val Val
260 265 270
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
275 280 285
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
290 295 300
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
305 310 315 320
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
370 375 380
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
385 390 395 400
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
405 410 415
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
420 425 430
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys
435 440 445
Arg Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
450 455 460
Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu
465 470 475 480
Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr
485 490 495
Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu
500 505 510
Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg
515 520 525
Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser
530 535 540
Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser
545 550 555 560
Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser
565 570 575
Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile
580 585 590
Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln
595 600 605
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
610 615 620
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
625 630 635 640
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
645 650 655
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
660 665 670
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
675 680 685
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Phe
690 695 700
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
705 710 715 720
Val Thr Val Ala Phe Ile Ile Phe Trp Val Lys Arg Gly Arg Lys Lys
725 730 735
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
740 745 750
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
755 760 765
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
770 775 780
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
785 790 795 800
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
805 810 815
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
820 825 830
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
835 840 845
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
850 855 860
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
865 870 875 880
Leu Pro Pro Arg
<210> 38
<211> 882
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 38
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Cys Asp Ile Tyr Ile Trp
260 265 270
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
275 280 285
Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
290 295 300
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
305 310 315 320
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys
325 330 335
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
340 345 350
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
355 360 365
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
370 375 380
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
385 390 395 400
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
405 410 415
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
420 425 430
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ala Lys Arg
435 440 445
Ser Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp
450 455 460
Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu Leu Gly Leu Leu Ile
465 470 475 480
Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys Gln Glu Val Thr Gln
485 490 495
Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu Asn Leu Val Leu Asn
500 505 510
Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu Gln Trp Phe Arg Gln
515 520 525
Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu Ile Gln Ser Ser Gln
530 535 540
Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser Leu Asp Lys Ser Ser
545 550 555 560
Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln Pro Gly Asp Ser Ala
565 570 575
Thr Tyr Leu Cys Ala Val Arg Pro Leu Tyr Gly Gly Ser Tyr Ile Pro
580 585 590
Thr Phe Gly Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln Asn
595 600 605
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
610 615 620
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
625 630 635 640
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met
645 650 655
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
660 665 670
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
675 680 685
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Gly Ser Pro Lys Cys Asp
690 695 700
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
705 710 715 720
Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu
725 730 735
Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu
740 745 750
Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys
755 760 765
Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
770 775 780
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
785 790 795 800
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
805 810 815
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
820 825 830
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
835 840 845
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
850 855 860
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
865 870 875 880
Pro Arg
<210> 39
<211> 965
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 39
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Cys Asp Ile Tyr Ile Trp
260 265 270
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
275 280 285
Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
290 295 300
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
305 310 315 320
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Ser Lys
325 330 335
Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg
340 345 350
Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp
355 360 365
Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg Ala Lys Arg Ser Gly Ser Gly Ala Thr Asn
485 490 495
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
500 505 510
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
515 520 525
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
530 535 540
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
545 550 555 560
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
565 570 575
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
580 585 590
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
595 600 605
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
610 615 620
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
625 630 635 640
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
645 650 655
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
660 665 670
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
675 680 685
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
690 695 700
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
705 710 715 720
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
725 730 735
Glu Ser Ser Gly Ser Pro Lys Cys Asp Ile Tyr Ile Trp Ala Pro Leu
740 745 750
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
755 760 765
Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
770 775 780
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
785 790 795 800
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Lys Arg Gly Arg Lys
805 810 815
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
820 825 830
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
835 840 845
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
850 855 860
Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
865 870 875 880
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
885 890 895
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
900 905 910
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
915 920 925
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
930 935 940
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
945 950 955 960
Ala Leu Pro Pro Arg
965
<210> 40
<211> 1059
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 40
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Ala Lys Pro Thr Thr Thr
260 265 270
Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro
275 280 285
Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val
290 295 300
His Thr Arg Gly Leu Asp Phe Ala Pro Arg Lys Ile Glu Val Met Tyr
305 310 315 320
Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn Gly Thr Ile Ile His
325 330 335
Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser
340 345 350
Lys Pro Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr
355 360 365
Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys
370 375 380
Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg
385 390 395 400
Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp
405 410 415
Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
420 425 430
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
435 440 445
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
450 455 460
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
465 470 475 480
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
485 490 495
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
500 505 510
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
515 520 525
Gln Ala Leu Pro Pro Arg Ala Lys Arg Ser Gly Ser Gly Ala Thr Asn
530 535 540
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
545 550 555 560
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
565 570 575
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
580 585 590
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
595 600 605
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
610 615 620
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
625 630 635 640
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
645 650 655
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
660 665 670
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
675 680 685
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
690 695 700
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
705 710 715 720
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
725 730 735
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
740 745 750
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
755 760 765
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
770 775 780
Glu Ser Ser Gly Ser Pro Lys Ala Lys Pro Thr Thr Thr Pro Ala Pro
785 790 795 800
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
805 810 815
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
820 825 830
Gly Leu Asp Phe Ala Pro Arg Lys Ile Glu Val Met Tyr Pro Pro Pro
835 840 845
Tyr Leu Asp Asn Glu Lys Ser Asn Gly Thr Ile Ile His Val Lys Gly
850 855 860
Lys His Leu Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser Lys Pro Phe
865 870 875 880
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
885 890 895
Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg
900 905 910
Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro
915 920 925
Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala
930 935 940
Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
945 950 955 960
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
965 970 975
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
980 985 990
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
995 1000 1005
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
1010 1015 1020
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
1025 1030 1035
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
1040 1045 1050
Gln Ala Leu Pro Pro Arg
1055
<210> 41
<211> 859
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 41
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Gly Ser Pro Lys Ala Lys Pro Thr Thr Thr
260 265 270
Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro
275 280 285
Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val
290 295 300
His Thr Arg Gly Leu Asp Phe Ala Pro Arg Lys Ile Glu Val Met Tyr
305 310 315 320
Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn Gly Thr Ile Ile His
325 330 335
Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser
340 345 350
Lys Pro Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr
355 360 365
Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys
370 375 380
Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg
385 390 395 400
Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp
405 410 415
Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
420 425 430
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
435 440 445
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
450 455 460
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
465 470 475 480
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
485 490 495
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
500 505 510
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
515 520 525
Gln Ala Leu Pro Pro Arg Ala Lys Arg Ser Gly Ser Gly Ala Thr Asn
530 535 540
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
545 550 555 560
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
565 570 575
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
580 585 590
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
595 600 605
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
610 615 620
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
625 630 635 640
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
645 650 655
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
660 665 670
Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser
675 680 685
Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
690 695 700
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
705 710 715 720
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
725 730 735
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
740 745 750
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
755 760 765
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
770 775 780
Glu Ser Ser Gly Ser Pro Lys Cys Asp Ile Tyr Ile Trp Ala Pro Leu
785 790 795 800
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
805 810 815
Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
820 825 830
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
835 840 845
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
850 855
<210> 42
<211> 120
<212> DNA
<213> 智人
<400> 42
aaaattgaag ttatgtatcc tcctccttac ctagacaatg agaagagcaa tggaaccatt 60
atccatgtga aagggaaaca cctttgtcca agtcccctat ttcccggacc ttctaagccc 120
<210> 43
<211> 120
<212> DNA
<213> 智人
<400> 43
aagatcgagg taatgtaccc accgccctat cttgataacg aaaaatctaa cggtacaata 60
attcacgtca agggcaagca tttgtgccct tccccgttgt tcccgggccc aagcaaaccg 120
<210> 44
<211> 81
<212> DNA
<213> 智人
<400> 44
ttttgggtgc tggtggtggt tggtggagtc ctggcttgct atagcttgct agtaacagtg 60
gcctttatta ttttctgggt g 81
<210> 45
<211> 81
<212> DNA
<213> 智人
<400> 45
ttctgggttc tcgtcgtcgt gggaggtgtg ttagcatgtt actctctctt ggttactgtc 60
gctttcataa tcttttgggt c 81
<210> 46
<211> 123
<212> DNA
<213> 智人
<400> 46
aggagtaaga ggagcaggct cctgcacagt gactacatga acatgactcc ccgccgcccc 60
gggcccaccc gcaagcatta ccagccctat gccccaccac gcgacttcgc agcctatcgc 120
tcc 123
<210> 47
<211> 123
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 47
cgctcaaaac gctctcgctt gttacattcc gattatatga atatgacacc taggagacct 60
ggcccgacta ggaaacacta tcaaccttac gcacctccca gagattttgc tgcttacagg 120
agt 123
<210> 48
<211> 126
<212> DNA
<213> 智人
<400> 48
aaacggggca gaaagaaact cctgtatata ttcaaacaac catttatgag accagtacaa 60
actactcaag aggaagatgg ctgtagctgc cgatttccag aagaagaaga aggaggatgt 120
gaactg 126
<210> 49
<211> 126
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 49
aagagagggc gtaaaaagct gctctacatc tttaagcagc ctttcatgcg tcctgttcag 60
acaacacagg aagaggacgg atgctcttgc aggttccctg aggaggagga gggtggttgc 120
gagctc 126
<210> 50
<211> 138
<212> DNA
<213> 智人
<400> 50
gctaagccca ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 60
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 120
agggggctgg acttcgcc 138
<210> 51
<211> 138
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 51
gctaagccca ctactacccc agctcccagg cctcccacac ctgccccaac aatcgccagc 60
cagccactgt cccttaggcc cgaggcctgt aggcccgccg ccggaggagc cgtgcacacc 120
cgcggactgg attttgct 138
<210> 52
<211> 78
<212> DNA
<213> 智人
<400> 52
tgtgatatct acatctgggc gcccttggcc gggacttgtg gggtccttct cctgtcactg 60
gttatcaccc tttactgc 78
<210> 53
<211> 78
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 53
tgcgacattt atatttgggc ccctctcgct ggcacatgcg gcgtgttgtt gctcagcctc 60
gtgattacac tttattgt 78
<210> 54
<211> 2652
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 54
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtg cgacatttat atttgggccc ctctcgctgg cacatgcggc 840
gtgttgttgc tcagcctcgt gattacactt tattgtaaga gagggcgtaa aaagctgctc 900
tacatcttta agcagccttt catgcgtcct gttcagacaa cacaggaaga ggacggatgc 960
tcttgcaggt tccctgagga ggaggagggt ggttgcgagc tccgggtcaa attttcacgc 1020
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1080
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1140
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1200
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1260
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1320
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1380
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1440
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1500
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1560
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1620
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1680
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1740
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1800
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1860
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 1920
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 1980
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2040
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2100
tccccaaaat gtgatatcta catctgggcg cccttggccg ggacttgtgg ggtccttctc 2160
ctgtcactgg ttatcaccct ttactgcaaa cggggcagaa agaaactcct gtatatattc 2220
aaacaaccat ttatgagacc agtacaaact actcaagagg aagatggctg tagctgccga 2280
tttccagaag aagaagaagg aggatgtgaa ctgagagtga agttcagcag gagcgcagac 2340
gcccccgcgt accagcaggg ccagaaccag ctctataacg agctcaatct aggacgaaga 2400
gaggagtacg atgttttgga caagagacgt ggccgggacc ctgagatggg gggaaagccg 2460
agaaggaaga accctcagga aggcctgtac aatgaactgc agaaagataa gatggcggag 2520
gcctacagtg agattgggat gaaaggcgag cgccggaggg gcaaggggca cgatggcctt 2580
taccagggtc tcagtacagc caccaaggac acctacgacg cccttcacat gcaggccctg 2640
ccccctcgct aa 2652
<210> 55
<211> 2658
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 55
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtca agagagggcg taaaaagctg 900
ctctacatct ttaagcagcc tttcatgcgt cctgttcaga caacacagga agaggacgga 960
tgctcttgca ggttccctga ggaggaggag ggtggttgcg agctccgggt caaattttca 1020
cgctccgctg atgctcctgc ctatcaacaa gggcaaaatc aattgtacaa tgaattgaac 1080
ttgggtagaa gggaagaata tgacgtgctc gataaacgga gggggagaga tccagaaatg 1140
ggcggtaaac cacggcgcaa aaatccacaa gagggattgt ataacgagct ccaaaaggac 1200
aaaatggcag aagcttattc agaaatagga atgaaggggg aaaggagacg aggtaaaggt 1260
catgacggat tgtatcaagg attgtcaacc gctactaaag atacatatga tgctttgcat 1320
atgcaagctt tgcctcccag agccaagcgg tctgggtctg gggccaccaa cttcagcctg 1380
ctgaagcagg ccggcgacgt ggaggagaac cccggcccca tggagaccct cttgggcctg 1440
cttatccttt ggctgcagct gcaatgggtg agcagcaaac aggaggtgac gcagattcct 1500
gcagctctga gtgtcccaga aggagaaaac ttggttctca actgcagttt cactgatagc 1560
gctatttaca acctccagtg gtttaggcag gaccctggga aaggtctcac atctctgttg 1620
cttattcagt caagtcagag agagcaaaca agtggaagac ttaatgcctc gctggataaa 1680
tcatcaggac gtagtacttt atacattgca gcttctcagc ctggtgactc agccacctac 1740
ctctgtgctg tgaggcccct gtacggagga agctacatac ctacatttgg aagaggaacc 1800
agccttattg ttcatccgta tatccagaac cctgaccctg ccgtgtacca gctgagagac 1860
tctaaatcca gtgacaagtc tgtctgccta ttcaccgatt ttgattctca aacaaatgtg 1920
tcacaaagta aggattctga tgtgtatatc acagacaaaa ctgtgctaga catgaggtct 1980
atggacttca agagcaacag tgctgtggcc tggagcaaca aatctgactt tgcatgtgca 2040
aacgccttca acaacagcat tattccagaa gacaccttct tccccagccc agaaagttcc 2100
ggctccccaa aattttgggt gctggtggtg gttggtggag tcctggcttg ctatagcttg 2160
ctagtaacag tggcctttat tattttctgg gtgaaacggg gcagaaagaa actcctgtat 2220
atattcaaac aaccatttat gagaccagta caaactactc aagaggaaga tggctgtagc 2280
tgccgatttc cagaagaaga agaaggagga tgtgaactga gagtgaagtt cagcaggagc 2340
gcagacgccc ccgcgtacca gcagggccag aaccagctct ataacgagct caatctagga 2400
cgaagagagg agtacgatgt tttggacaag agacgtggcc gggaccctga gatgggggga 2460
aagccgagaa ggaagaaccc tcaggaaggc ctgtacaatg aactgcagaa agataagatg 2520
gcggaggcct acagtgagat tgggatgaaa ggcgagcgcc ggaggggcaa ggggcacgat 2580
ggcctttacc agggtctcag tacagccacc aaggacacct acgacgccct tcacatgcag 2640
gccctgcccc ctcgctaa 2658
<210> 56
<211> 2904
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 56
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtaagagagg gcgtaaaaag 1020
ctgctctaca tctttaagca gcctttcatg cgtcctgttc agacaacaca ggaagaggac 1080
ggatgctctt gcaggttccc tgaggaggag gagggtggtt gcgagctccg ggtcaaattt 1140
tcacgctccg ctgatgctcc tgcctatcaa caagggcaaa atcaattgta caatgaattg 1200
aacttgggta gaagggaaga atatgacgtg ctcgataaac ggagggggag agatccagaa 1260
atgggcggta aaccacggcg caaaaatcca caagagggat tgtataacga gctccaaaag 1320
gacaaaatgg cagaagctta ttcagaaata ggaatgaagg gggaaaggag acgaggtaaa 1380
ggtcatgacg gattgtatca aggattgtca accgctacta aagatacata tgatgctttg 1440
catatgcaag ctttgcctcc cagagccaag cggtctgggt ctggggccac caacttcagc 1500
ctgctgaagc aggccggcga cgtggaggag aaccccggcc ccatggagac cctcttgggc 1560
ctgcttatcc tttggctgca gctgcaatgg gtgagcagca aacaggaggt gacgcagatt 1620
cctgcagctc tgagtgtccc agaaggagaa aacttggttc tcaactgcag tttcactgat 1680
agcgctattt acaacctcca gtggtttagg caggaccctg ggaaaggtct cacatctctg 1740
ttgcttattc agtcaagtca gagagagcaa acaagtggaa gacttaatgc ctcgctggat 1800
aaatcatcag gacgtagtac tttatacatt gcagcttctc agcctggtga ctcagccacc 1860
tacctctgtg ctgtgaggcc cctgtacgga ggaagctaca tacctacatt tggaagagga 1920
accagcctta ttgttcatcc gtatatccag aaccctgacc ctgccgtgta ccagctgaga 1980
gactctaaat ccagtgacaa gtctgtctgc ctattcaccg attttgattc tcaaacaaat 2040
gtgtcacaaa gtaaggattc tgatgtgtat atcacagaca aaactgtgct agacatgagg 2100
tctatggact tcaagagcaa cagtgctgtg gcctggagca acaaatctga ctttgcatgt 2160
gcaaacgcct tcaacaacag cattattcca gaagacacct tcttccccag cccagaaagt 2220
tccggctccc caaaattttg ggtgctggtg gtggttggtg gagtcctggc ttgctatagc 2280
ttgctagtaa cagtggcctt tattattttc tgggtgagga gtaagaggag caggctcctg 2340
cacagtgact acatgaacat gactccccgc cgccccgggc ccacccgcaa gcattaccag 2400
ccctatgccc caccacgcga cttcgcagcc tatcgctcca aacggggcag aaagaaactc 2460
ctgtatatat tcaaacaacc atttatgaga ccagtacaaa ctactcaaga ggaagatggc 2520
tgtagctgcc gatttccaga agaagaagaa ggaggatgtg aactgagagt gaagttcagc 2580
aggagcgcag acgcccccgc gtaccagcag ggccagaacc agctctataa cgagctcaat 2640
ctaggacgaa gagaggagta cgatgttttg gacaagagac gtggccggga ccctgagatg 2700
gggggaaagc cgagaaggaa gaaccctcag gaaggcctgt acaatgaact gcagaaagat 2760
aagatggcgg aggcctacag tgagattggg atgaaaggcg agcgccggag gggcaagggg 2820
cacgatggcc tttaccaggg tctcagtaca gccaccaagg acacctacga cgcccttcac 2880
atgcaggccc tgccccctcg ctaa 2904
<210> 57
<211> 2652
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 57
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtcgggtcaa attttcacgc 1020
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1080
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1140
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1200
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1260
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1320
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1380
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1440
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1500
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1560
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1620
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1680
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1740
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1800
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1860
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 1920
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 1980
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2040
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2100
tccccaaaat gtgatatcta catctgggcg cccttggccg ggacttgtgg ggtccttctc 2160
ctgtcactgg ttatcaccct ttactgcaaa cggggcagaa agaaactcct gtatatattc 2220
aaacaaccat ttatgagacc agtacaaact actcaagagg aagatggctg tagctgccga 2280
tttccagaag aagaagaagg aggatgtgaa ctgagagtga agttcagcag gagcgcagac 2340
gcccccgcgt accagcaggg ccagaaccag ctctataacg agctcaatct aggacgaaga 2400
gaggagtacg atgttttgga caagagacgt ggccgggacc ctgagatggg gggaaagccg 2460
agaaggaaga accctcagga aggcctgtac aatgaactgc agaaagataa gatggcggag 2520
gcctacagtg agattgggat gaaaggcgag cgccggaggg gcaaggggca cgatggcctt 2580
taccagggtc tcagtacagc caccaaggac acctacgacg cccttcacat gcaggccctg 2640
ccccctcgct aa 2652
<210> 58
<211> 2316
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 58
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtcgggtcaa attttcacgc 1020
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1080
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1140
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1200
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1260
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1320
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1380
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1440
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1500
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1560
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1620
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1680
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1740
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1800
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1860
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 1920
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 1980
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2040
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2100
tccccaaaat gtgatatcta catctgggcg cccttggccg ggacttgtgg ggtccttctc 2160
ctgtcactgg ttatcaccct ttactgcaaa cggggcagaa agaaactcct gtatatattc 2220
aaacaaccat ttatgagacc agtacaaact actcaagagg aagatggctg tagctgccga 2280
tttccagaag aagaagaagg aggatgtgaa ctgtaa 2316
<210> 59
<211> 2655
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 59
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtt ctgggttctc gtcgtcgtgg gaggtgtgtt agcatgttac 840
tctctcttgg ttactgtcgc tttcataatc ttttgggtcc gctcaaaacg ctctcgcttg 900
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 960
caaccttacg cacctcccag agattttgct gcttacagga gtcgggtcaa attttcacgc 1020
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1080
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1140
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1200
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1260
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1320
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1380
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1440
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1500
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1560
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1620
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1680
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1740
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1800
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1860
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 1920
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 1980
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2040
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2100
tccccaaaat tttgggtgct ggtggtggtt ggtggagtcc tggcttgcta tagcttgcta 2160
gtaacagtgg cctttattat tttctgggtg aaacggggca gaaagaaact cctgtatata 2220
ttcaaacaac catttatgag accagtacaa actactcaag aggaagatgg ctgtagctgc 2280
cgatttccag aagaagaaga aggaggatgt gaactgagag tgaagttcag caggagcgca 2340
gacgcccccg cgtaccagca gggccagaac cagctctata acgagctcaa tctaggacga 2400
agagaggagt acgatgtttt ggacaagaga cgtggccggg accctgagat ggggggaaag 2460
ccgagaagga agaaccctca ggaaggcctg tacaatgaac tgcagaaaga taagatggcg 2520
gaggcctaca gtgagattgg gatgaaaggc gagcgccgga ggggcaaggg gcacgatggc 2580
ctttaccagg gtctcagtac agccaccaag gacacctacg acgcccttca catgcaggcc 2640
ctgccccctc gctaa 2655
<210> 60
<211> 2649
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 60
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtg cgacatttat atttgggccc ctctcgctgg cacatgcggc 840
gtgttgttgc tcagcctcgt gattacactt tattgtcgct caaaacgctc tcgcttgtta 900
cattccgatt atatgaatat gacacctagg agacctggcc cgactaggaa acactatcaa 960
ccttacgcac ctcccagaga ttttgctgct tacaggagtc gggtcaaatt ttcacgctcc 1020
gctgatgctc ctgcctatca acaagggcaa aatcaattgt acaatgaatt gaacttgggt 1080
agaagggaag aatatgacgt gctcgataaa cggaggggga gagatccaga aatgggcggt 1140
aaaccacggc gcaaaaatcc acaagaggga ttgtataacg agctccaaaa ggacaaaatg 1200
gcagaagctt attcagaaat aggaatgaag ggggaaagga gacgaggtaa aggtcatgac 1260
ggattgtatc aaggattgtc aaccgctact aaagatacat atgatgcttt gcatatgcaa 1320
gctttgcctc ccagagccaa gcggtctggg tctggggcca ccaacttcag cctgctgaag 1380
caggccggcg acgtggagga gaaccccggc cccatggaga ccctcttggg cctgcttatc 1440
ctttggctgc agctgcaatg ggtgagcagc aaacaggagg tgacgcagat tcctgcagct 1500
ctgagtgtcc cagaaggaga aaacttggtt ctcaactgca gtttcactga tagcgctatt 1560
tacaacctcc agtggtttag gcaggaccct gggaaaggtc tcacatctct gttgcttatt 1620
cagtcaagtc agagagagca aacaagtgga agacttaatg cctcgctgga taaatcatca 1680
ggacgtagta ctttatacat tgcagcttct cagcctggtg actcagccac ctacctctgt 1740
gctgtgaggc ccctgtacgg aggaagctac atacctacat ttggaagagg aaccagcctt 1800
attgttcatc cgtatatcca gaaccctgac cctgccgtgt accagctgag agactctaaa 1860
tccagtgaca agtctgtctg cctattcacc gattttgatt ctcaaacaaa tgtgtcacaa 1920
agtaaggatt ctgatgtgta tatcacagac aaaactgtgc tagacatgag gtctatggac 1980
ttcaagagca acagtgctgt ggcctggagc aacaaatctg actttgcatg tgcaaacgcc 2040
ttcaacaaca gcattattcc agaagacacc ttcttcccca gcccagaaag ttccggctcc 2100
ccaaaatgtg atatctacat ctgggcgccc ttggccggga cttgtggggt ccttctcctg 2160
tcactggtta tcacccttta ctgcaaacgg ggcagaaaga aactcctgta tatattcaaa 2220
caaccattta tgagaccagt acaaactact caagaggaag atggctgtag ctgccgattt 2280
ccagaagaag aagaaggagg atgtgaactg agagtgaagt tcagcaggag cgcagacgcc 2340
cccgcgtacc agcagggcca gaaccagctc tataacgagc tcaatctagg acgaagagag 2400
gagtacgatg ttttggacaa gagacgtggc cgggaccctg agatgggggg aaagccgaga 2460
aggaagaacc ctcaggaagg cctgtacaat gaactgcaga aagataagat ggcggaggcc 2520
tacagtgaga ttgggatgaa aggcgagcgc cggaggggca aggggcacga tggcctttac 2580
cagggtctca gtacagccac caaggacacc tacgacgccc ttcacatgca ggccctgccc 2640
cctcgctaa 2649
<210> 61
<211> 2898
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 61
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaagtg cgacatttat atttgggccc ctctcgctgg cacatgcggc 840
gtgttgttgc tcagcctcgt gattacactt tattgtagga gtaagaggag caggctcctg 900
cacagtgact acatgaacat gactccccgc cgccccgggc ccacccgcaa gcattaccag 960
ccctatgccc caccacgcga cttcgcagcc tatcgctccc gctcaaaacg ctctcgcttg 1020
ttacattccg attatatgaa tatgacacct aggagacctg gcccgactag gaaacactat 1080
caaccttacg cacctcccag agattttgct gcttacagga gtcgggtcaa attttcacgc 1140
tccgctgatg ctcctgccta tcaacaaggg caaaatcaat tgtacaatga attgaacttg 1200
ggtagaaggg aagaatatga cgtgctcgat aaacggaggg ggagagatcc agaaatgggc 1260
ggtaaaccac ggcgcaaaaa tccacaagag ggattgtata acgagctcca aaaggacaaa 1320
atggcagaag cttattcaga aataggaatg aagggggaaa ggagacgagg taaaggtcat 1380
gacggattgt atcaaggatt gtcaaccgct actaaagata catatgatgc tttgcatatg 1440
caagctttgc ctcccagagc caagcggtct gggtctgggg ccaccaactt cagcctgctg 1500
aagcaggccg gcgacgtgga ggagaacccc ggccccatgg agaccctctt gggcctgctt 1560
atcctttggc tgcagctgca atgggtgagc agcaaacagg aggtgacgca gattcctgca 1620
gctctgagtg tcccagaagg agaaaacttg gttctcaact gcagtttcac tgatagcgct 1680
atttacaacc tccagtggtt taggcaggac cctgggaaag gtctcacatc tctgttgctt 1740
attcagtcaa gtcagagaga gcaaacaagt ggaagactta atgcctcgct ggataaatca 1800
tcaggacgta gtactttata cattgcagct tctcagcctg gtgactcagc cacctacctc 1860
tgtgctgtga ggcccctgta cggaggaagc tacataccta catttggaag aggaaccagc 1920
cttattgttc atccgtatat ccagaaccct gaccctgccg tgtaccagct gagagactct 1980
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 2040
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 2100
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2160
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccaga aagttccggc 2220
tccccaaaat gtgatatcta catctgggcg cccttggccg ggacttgtgg ggtccttctc 2280
ctgtcactgg ttatcaccct ttactgcaag agagggcgta aaaagctgct ctacatcttt 2340
aagcagcctt tcatgcgtcc tgttcagaca acacaggaag aggacggatg ctcttgcagg 2400
ttccctgagg aggaggaggg tggttgcgag ctcaaacggg gcagaaagaa actcctgtat 2460
atattcaaac aaccatttat gagaccagta caaactactc aagaggaaga tggctgtagc 2520
tgccgatttc cagaagaaga agaaggagga tgtgaactga gagtgaagtt cagcaggagc 2580
gcagacgccc ccgcgtacca gcagggccag aaccagctct ataacgagct caatctagga 2640
cgaagagagg agtacgatgt tttggacaag agacgtggcc gggaccctga gatgggggga 2700
aagccgagaa ggaagaaccc tcaggaaggc ctgtacaatg aactgcagaa agataagatg 2760
gcggaggcct acagtgagat tgggatgaaa ggcgagcgcc ggaggggcaa ggggcacgat 2820
ggcctttacc agggtctcag tacagccacc aaggacacct acgacgccct tcacatgcag 2880
gccctgcccc ctcgctaa 2898
<210> 62
<211> 3180
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 62
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaaggc aaaaccgacg accacccctg cccccaggcc tcctactccc 840
gccccgacga ttgccagcca accgttaagt ttaagaccgg aagcatgtag accggcagct 900
ggtggggctg ttcatacacg tggcttagat tttgcgccta ggaagatcga ggtaatgtac 960
ccaccgccct atcttgataa cgaaaaatct aacggtacaa taattcacgt caagggcaag 1020
catttgtgcc cttccccgtt gttcccgggc ccaagcaaac cgttctgggt tctcgtcgtc 1080
gtgggaggtg tgttagcatg ttactctctc ttggttactg tcgctttcat aatcttttgg 1140
gtccgctcaa aacgctctcg cttgttacat tccgattata tgaatatgac acctaggaga 1200
cctggcccga ctaggaaaca ctatcaacct tacgcacctc ccagagattt tgctgcttac 1260
aggagtcggg tcaaattttc acgctccgct gatgctcctg cctatcaaca agggcaaaat 1320
caattgtaca atgaattgaa cttgggtaga agggaagaat atgacgtgct cgataaacgg 1380
agggggagag atccagaaat gggcggtaaa ccacggcgca aaaatccaca agagggattg 1440
tataacgagc tccaaaagga caaaatggca gaagcttatt cagaaatagg aatgaagggg 1500
gaaaggagac gaggtaaagg tcatgacgga ttgtatcaag gattgtcaac cgctactaaa 1560
gatacatatg atgctttgca tatgcaagct ttgcctccca gagccaagcg gtctgggtct 1620
ggggccacca acttcagcct gctgaagcag gccggcgacg tggaggagaa ccccggcccc 1680
atggagaccc tcttgggcct gcttatcctt tggctgcagc tgcaatgggt gagcagcaaa 1740
caggaggtga cgcagattcc tgcagctctg agtgtcccag aaggagaaaa cttggttctc 1800
aactgcagtt tcactgatag cgctatttac aacctccagt ggtttaggca ggaccctggg 1860
aaaggtctca catctctgtt gcttattcag tcaagtcaga gagagcaaac aagtggaaga 1920
cttaatgcct cgctggataa atcatcagga cgtagtactt tatacattgc agcttctcag 1980
cctggtgact cagccaccta cctctgtgct gtgaggcccc tgtacggagg aagctacata 2040
cctacatttg gaagaggaac cagccttatt gttcatccgt atatccagaa ccctgaccct 2100
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 2160
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 2220
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 2280
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 2340
ttccccagcc cagaaagttc cggctcccca aaagctaagc ccaccacgac gccagcgccg 2400
cgaccaccaa caccggcgcc caccatcgcg tcgcagcccc tgtccctgcg cccagaggcg 2460
tgccggccag cggcgggggg cgcagtgcac acgagggggc tggacttcgc ccctaggaaa 2520
attgaagtta tgtatcctcc tccttaccta gacaatgaga agagcaatgg aaccattatc 2580
catgtgaaag ggaaacacct ttgtccaagt cccctatttc ccggaccttc taagcccttt 2640
tgggtgctgg tggtggttgg tggagtcctg gcttgctata gcttgctagt aacagtggcc 2700
tttattattt tctgggtgag gagtaagagg agcaggctcc tgcacagtga ctacatgaac 2760
atgactcccc gccgccccgg gcccacccgc aagcattacc agccctatgc cccaccacgc 2820
gacttcgcag cctatcgctc cagagtgaag ttcagcagga gcgcagacgc ccccgcgtac 2880
cagcagggcc agaaccagct ctataacgag ctcaatctag gacgaagaga ggagtacgat 2940
gttttggaca agagacgtgg ccgggaccct gagatggggg gaaagccgag aaggaagaac 3000
cctcaggaag gcctgtacaa tgaactgcag aaagataaga tggcggaggc ctacagtgag 3060
attgggatga aaggcgagcg ccggaggggc aaggggcacg atggccttta ccagggtctc 3120
agtacagcca ccaaggacac ctacgacgcc cttcacatgc aggccctgcc ccctcgctaa 3180
<210> 63
<211> 2580
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 63
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagacggct ctcctaaggc aaaaccgacg accacccctg cccccaggcc tcctactccc 840
gccccgacga ttgccagcca accgttaagt ttaagaccgg aagcatgtag accggcagct 900
ggtggggctg ttcatacacg tggcttagat tttgcgccta ggaagatcga ggtaatgtac 960
ccaccgccct atcttgataa cgaaaaatct aacggtacaa taattcacgt caagggcaag 1020
catttgtgcc cttccccgtt gttcccgggc ccaagcaaac cgttctgggt tctcgtcgtc 1080
gtgggaggtg tgttagcatg ttactctctc ttggttactg tcgctttcat aatcttttgg 1140
gtccgctcaa aacgctctcg cttgttacat tccgattata tgaatatgac acctaggaga 1200
cctggcccga ctaggaaaca ctatcaacct tacgcacctc ccagagattt tgctgcttac 1260
aggagtcggg tcaaattttc acgctccgct gatgctcctg cctatcaaca agggcaaaat 1320
caattgtaca atgaattgaa cttgggtaga agggaagaat atgacgtgct cgataaacgg 1380
agggggagag atccagaaat gggcggtaaa ccacggcgca aaaatccaca agagggattg 1440
tataacgagc tccaaaagga caaaatggca gaagcttatt cagaaatagg aatgaagggg 1500
gaaaggagac gaggtaaagg tcatgacgga ttgtatcaag gattgtcaac cgctactaaa 1560
gatacatatg atgctttgca tatgcaagct ttgcctccca gagccaagcg gtctgggtct 1620
ggggccacca acttcagcct gctgaagcag gccggcgacg tggaggagaa ccccggcccc 1680
atggagaccc tcttgggcct gcttatcctt tggctgcagc tgcaatgggt gagcagcaaa 1740
caggaggtga cgcagattcc tgcagctctg agtgtcccag aaggagaaaa cttggttctc 1800
aactgcagtt tcactgatag cgctatttac aacctccagt ggtttaggca ggaccctggg 1860
aaaggtctca catctctgtt gcttattcag tcaagtcaga gagagcaaac aagtggaaga 1920
cttaatgcct cgctggataa atcatcagga cgtagtactt tatacattgc agcttctcag 1980
cctggtgact cagccaccta cctctgtgct gtgaggcccc tgtacggagg aagctacata 2040
cctacatttg gaagaggaac cagccttatt gttcatccgt atatccagaa ccctgaccct 2100
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 2160
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 2220
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 2280
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 2340
ttccccagcc cagaaagttc cggctcccca aaatgtgata tctacatctg ggcgcccttg 2400
gccgggactt gtggggtcct tctcctgtca ctggttatca ccctttactg caaacggggc 2460
agaaagaaac tcctgtatat attcaaacaa ccatttatga gaccagtaca aactactcaa 2520
gaggaagatg gctgtagctg ccgatttcca gaagaagaag aaggaggatg tgaactgtaa 2580
<210> 64
<211> 612
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 64
Met Ser Ile Gly Leu Leu Cys Cys Ala Ala Leu Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Ser Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Ala Gly Ile Thr Asp Gln Gly Glu Val Pro
65 70 75 80
Asn Gly Tyr Asn Val Ser Arg Ser Thr Thr Glu Asp Phe Pro Leu Arg
85 90 95
Leu Leu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Val Gly Asn Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Ala Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asp Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Ala Lys Arg Ser Gly Ser Gly Ala Thr
305 310 315 320
Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly
325 330 335
Pro Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln
340 345 350
Trp Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser
355 360 365
Val Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser
370 375 380
Ala Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu
385 390 395 400
Thr Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly
405 410 415
Arg Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr
420 425 430
Ile Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val
435 440 445
Arg Pro Leu Tyr Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr
450 455 460
Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser Arg
610
<210> 65
<211> 1839
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 65
atgagcatcg gcctcctgtg ctgtgcagcc ttgtctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccag gtcctgaaga caggacagag catgacactg 120
cagtgtgccc aggatatgaa ccatgaatac atgtcctggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gctggtatca ctgaccaagg agaagtcccc 240
aatggctaca atgtctccag atcaaccaca gaggatttcc cgctcaggct gctgtcggct 300
gctccctccc agacatctgt gtacttctgt gccagcagtt acgtcgggaa caccggggag 360
ctgttttttg gagaaggctc taggctgacc gtactggagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatacgc tctgagcagc cgcctgaggg tctcggccac cttctggcag 660
gacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggctatgg tcaagagaaa ggattccaga ggcgccaagc ggtctgggtc tggggccacc 960
aacttcagcc tgctgaagca ggccggcgac gtggaggaga accccggccc catggagacc 1020
ctcttgggcc tgcttatcct ttggctgcag ctgcaatggg tgagcagcaa acaggaggtg 1080
acgcagattc ctgcagctct gagtgtccca gaaggagaaa acttggttct caactgcagt 1140
ttcactgata gcgctattta caacctccag tggtttaggc aggaccctgg gaaaggtctc 1200
acatctctgt tgcttattca gtcaagtcag agagagcaaa caagtggaag acttaatgcc 1260
tcgctggata aatcatcagg acgtagtact ttatacattg cagcttctca gcctggtgac 1320
tcagccacct acctctgtgc tgtgaggccc ctgtacggag gaagctacat acctacattt 1380
ggaagaggaa ccagccttat tgttcatccg tatatccaga accctgaccc tgccgtgtac 1440
cagctgagag actctaaatc cagtgacaag tctgtctgcc tattcaccga ttttgattct 1500
caaacaaatg tgtcacaaag taaggattct gatgtgtata tcacagacaa aactgtgcta 1560
gacatgaggt ctatggactt caagagcaac agtgctgtgg cctggagcaa caaatctgac 1620
tttgcatgtg caaacgcctt caacaacagc attattccag aagacacctt cttccccagc 1680
ccagaaagtt cctgtgatgt caagctggtc gagaaaagct ttgaaacaga tacgaaccta 1740
aactttcaaa acctgtcagt gattgggttc cgaatcctcc tcctgaaagt ggccgggttt 1800
aatctgctca tgacgctgcg gctgtggtcc agccggtaa 1839
<210> 66
<211> 48
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 66
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
1 5 10 15
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
20 25 30
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser Arg
35 40 45
<210> 67
<211> 7
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 67
Ala Lys Arg Ser Gly Ser Gly
1 5
<210> 68
<211> 49
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 68
Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser Ala Thr
1 5 10 15
Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu
20 25 30
Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp Ser Arg
35 40 45
Gly
<210> 69
<211> 4
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 69
Gly Ser Pro Lys
1
<210> 70
<211> 2
<212> PRT
<213> 人工序列
<220>
<223> 合成的
<400> 70
Pro Arg
1
<210> 71
<211> 6
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 71
ctcgag 6
<210> 72
<211> 14
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 72
cagccagcgg ccgc 14
<210> 73
<211> 8
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 73
gcggccgc 8
- 上一篇:一种医用注射器针头装配设备
- 下一篇:具有增强的抗体导向免疫应答的人自然杀伤细胞亚群
